BED文件也有可能是1-based坐标系?
众所周知,描述基因组中位置信息的坐标系通常有1-based和0-based之分。举个例子,要表示某条序列的前5个碱基,如下图的ATGCA,用1-based坐标系就应该是1-5,而0-based坐标系应该是0-5(类似与Python中的切片):
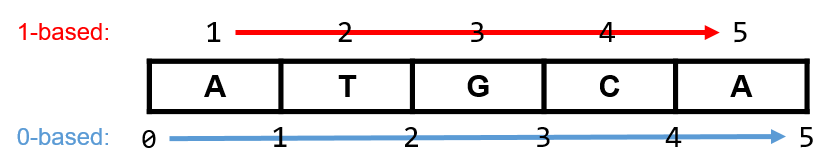
常见的1-based坐标系的有SAM、GFF/GTF、VCF、WIG、Axt、BLAST比对结果;0-based坐标系的文件有BAM、PSL、MAF(这里指的Multiple Alignment Format)、以及BED等。如果坐标区间范围很大,只做一些简单分析或者可视化,这一个碱基的增减可能影响不大。如果区间很小,比如TSS(Transcription Start Site,转录起始位点);又或者是要根据编码基因coding region的DNA序列转换成对应蛋白的氨基酸序列,那这一个碱基的差别可能就会带来严重错误。
上述这些0-based或者1-based坐标系的文件除了BED以外,基本由软件生成,出错的概率比较低,也就无需过于担心。而BED格式很多时候会通过custom script生成或者转换自其他格式,这就很容易出错了。虽然稍微花点心思就能查到UCSC Genome Browser FAQ中对BED等文件格式的详细解释,网上也有不少中文资料,但我以前观察到公司给新员工出的编程考核题中,GFF转BED仍是一个出错的重灾区,连不少工作几年的老员工都可能不清楚。已发表的论文中这些情况也不胜枚举,甚至最近发现连著名的FANTOM5 Project中激活增强子的BED文件也存在这个问题。下面是该文件前5行:
chr1 839741 840250 chr1:839741-840250 24 . 839787 839788 0,0,0 2 20,436 0,73
chr1 840753 841210 chr1:840753-841210 32 . 840811 840812 0,0,0 2 8,347 0,110
chr1 845485 845678 chr1:845485-845678 2 . 845581 845582 0,0,0 2 1,1 0,192
chr1 855764 856157 chr1:855764-856157 2 . 855855 855856 0,0,0 2 1,210 0,183
chr1 856539 856757 chr1:856539-856757 2 . 856713 856714 0,0,0 2 146,15 0,203根据UCSC的博文《The UCSC Genome Browser Coordinate Counting Systems》,BED文件第二列是0-based坐标系,而以chr#:##-##这样的“Position format”表示时,使用1-based坐标系,说明上面FANTOM5的这个BED文件,第二列和第四列中必有一处是错的。
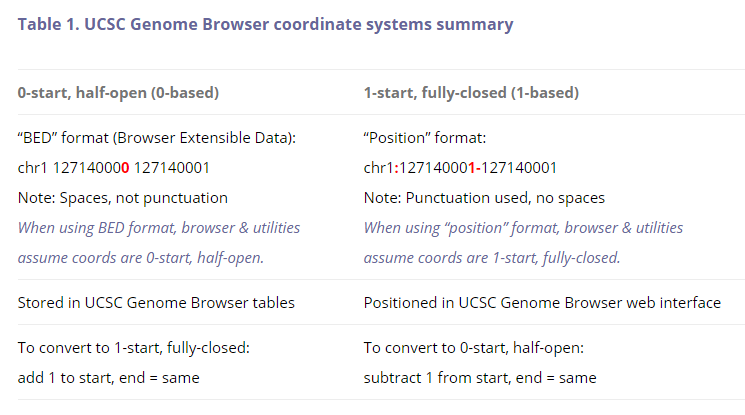
所以以后做分析,除了防止自己出错以外,可能还得推理一下别人给的BED文件是不是有问题,难受想哭。IGV默认根据读入文件的扩展名按照UCSC的坐标系标准进行处理,但似乎也因为错误的BED文件有点无奈——所以在track line中弄了个coords字段可以手动指定0-based坐标系或者1-based坐标系:
| Specifier | Value | Description |
|---|---|---|
| coords (IGV extension) | 0 / 1 | Indicate whether the file uses 0 or 1 based coordinates. The UCSC specification for WIG files uses 1 based coordinates and for BED files uses 0 based coordinates. If data looks off by one, check for a possible 0 vs 1 based coordinate issue. |
一个题外话:因为平时没有直接处理过二进制的BAM,都是通过samtools view转成SAM格式看的,所以BAM文件是0-based坐标系我也是最近才知道,甚至还小小震惊了一把 : )
 作者:
作者:
[...]利用参考基因组进行scaffolding提高组装连续性的工具整理 | Public Library of Bioinformatics MathJax.Hub.Config({showProcessingMessages:!1,messageStyle:"none",extensions:["tex2jax.js"],jax:["input/TeX","output/HTML-CSS"],[...]
sam和bam不一致是最逆天的,用pysam读取sam文件的时候,显示的比对起点和终点刚好是sam文件-1,自动和python下标的index计算方式一致了,当时我还以为是python的包故意调节的。
确实,pysam的position信息和SAM的相比都是-1的