HapHiC的适用范围
(更新时间:2023-11-26)
物种、分类群
我们的论文在动植物中对HapHiC进行了测试和验证,列表如下:
| 学名 | 俗名 | 倍性 | 单倍体分型 |
|---|---|---|---|
| Solanum tuberosum C88 | potato (马铃薯) | 同源四倍体 | 是 |
| Saccharum spontaneum Np-X | wild sugarcane (甘蔗) | 同源四倍体 | 是 |
| Saccharum spontaneum AP85-441 | wild sugarcane (甘蔗) | 同源四倍体 | 是 |
| Medicago sativa XinJiangDaYe | alfalfa (紫花苜蓿) | 同源四倍体 | 是 |
| Medicago sativa Zhongmu-4 | alfalfa (紫花苜蓿) | 同源四倍体 | 是 |
| Camellia sinensis Tieguanyin | Tea plant (茶树) | 二倍体 | 是 |
| Oryza alta | wild allotetraploid rice (水稻) | 异源四倍体 | 否 |
| Brassica napus | rapeseed (油菜) | 异源四倍体 | 否 |
| Eragrostis tef | teff (苔麸) | 异源四倍体 | 否 |
| Gossypium hirsutum | upland cotton (陆地棉) | 异源四倍体 | 否 |
| Triticum aestivum | bread wheat (小麦) | 异源六倍体 | 否 |
| Homo sapiens CHM13 | human (人) | 单倍体 | 否 |
| Oryza sativa | rice (水稻) | 二倍体 | 否 |
| Arabidopsis thaliana | thale cress (拟南芥) | 二倍体 | 否 |
| Ginkgo biloba | maidenhair tree (银杏) | 二倍体 | 否 |
| Corylus mandshurica | hairy hazel (毛榛) | 二倍体 | 否 |
| Papaver somniferum | opium poppy (罂粟) | 二倍体 | 否 |
| Camellia sinensis Huangdan | Tea plant (茶树) | 二倍体 | 否 |
| Echinochloa haploclada | barnyard grass (稗草) | 二倍体 | 否 |
| Prunus avium | sweet cherry (樱桃) | 二倍体 | 否 |
| Accipiter gentilis | Northern goshawk (苍鹰) | 二倍体 | 否 |
| Xenopus tropicalis | tropical clawed frog (热带爪蟾) | 二倍体 | 否 |
| Symphodus melops | corkwing wrasse (娇扁隆头鱼) | 二倍体 | 否 |
| Antheraea pernyi | Chinese oak silkmoth (柞蚕) | 二倍体 | 否 |
| Steromphala cineraria | gray topshell (螺) | 二倍体 | 否 |
| Lumbricus rubellus | humus earthworm (蚯蚓) | 二倍体 | 否 |
此外,一些用户也在其他高等植物、鱼类、寄生虫中使用了HapHiC并有不错的表现。
其他物种已知的一些情况:
- 在鸟类、爬行动物基因组中存在微染色体,这可能使得HapHiC在初步聚类过程中自动调参效果不佳。调参建议:可尝试分步运行HapHiC的流程,并在contig初步聚类过程中选择更高的
inflation值的聚类结果;在contig重新分配中使用更低的--min_group_len,防止微染色体的片段被过滤;此外,如果contig在各染色体中的长度分布不均匀(常见于contig N50较高时),考虑在contig初步聚类时使用更高的--Nx值,防止来源于微染色体的contig被过滤。 - 在真菌中,我们未做尝试,但是根据真菌基因组的大小,以及一些发表论文中真菌基因组Hi-C互作热图,我们推测contig的聚类可能会存在一些问题。并且由于每条染色体很短,在contig重新分配中需要使用更低的
--min_group_len。如果聚类出现问题,您还可以尝试不聚类直接进行排序的方法:quick view模式。至于细菌,我们也未进行尝试,但绝大多数细菌通过三代单分子测序可以直接组装成完成图,通常不需要进行挂载。
倍性、单倍体分型和杂合度
二倍体和多倍体基因组分型挂载的难点体现在多个方面。
其中一个是组装错误。分型基因组的组装过程更容易产生组装错误。HapHiC在预处理过程中通过一系列创新的方法尽可能地降低组装错误对contig的染色体分配的影响。这使得HapHiC可以在无参考基因组指导的情况下实现对二倍体和多倍体基因组的分型scaffolding。在模拟单倍体到同源十六倍体分型的测试中,HapHiC相较其他Hi-C scaffolding工具展现出明显更高的连续性和更低的染色体错误分配率。此外,我们也在多个同源四倍体植物和二倍体植物的分型组装中进行了进一步验证。
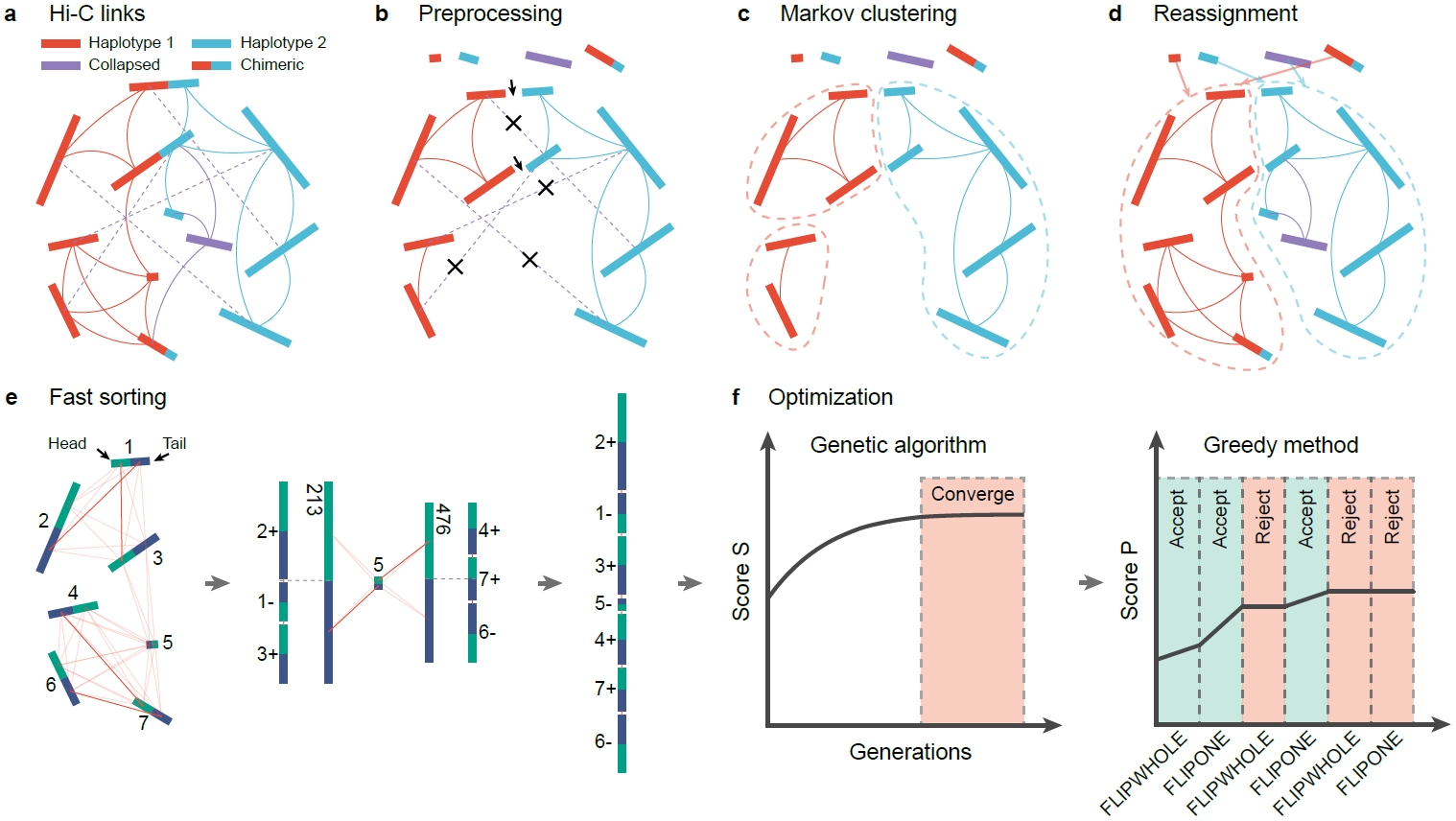
另一个难点是低杂合度。正如我们文章中提到的,只要变异在染色体上分布较为均匀,同源染色体之间极低的序列差异(即:低杂合度)对contig的染色体分配影响并不大。其主要影响是减少了有效Hi-C数据量(非特异性比对会被MAPQ过滤)。但有效Hi-C数据量下降对contig的排序会有一定程度的影响。一个很直观的反映就是在用Juicebox进行可视化的时候会因为热图颜色很浅甚至缺失数据感到无从下手。
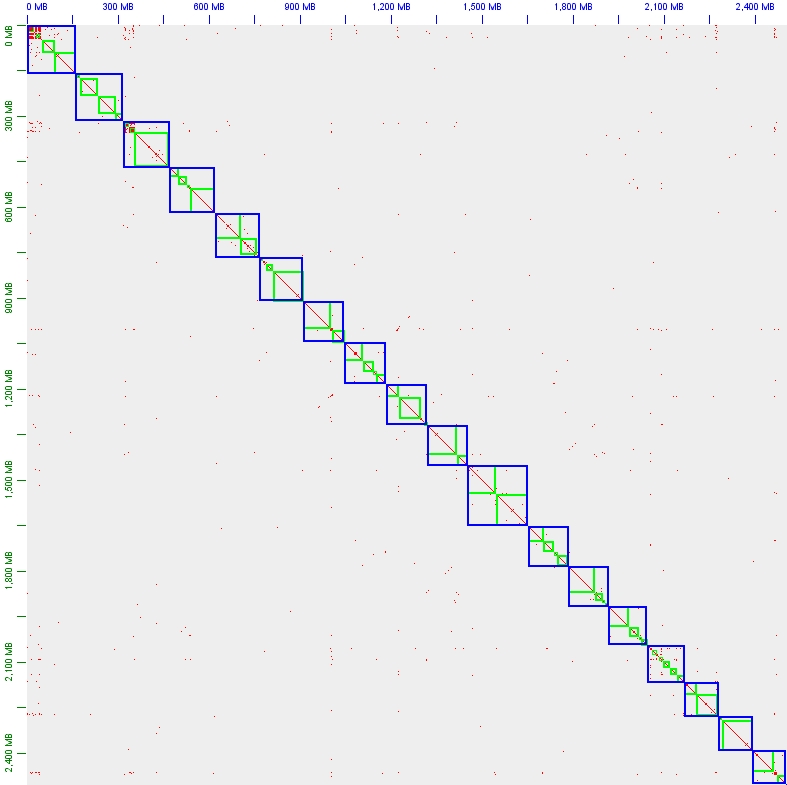
此外,单倍体之间的变异在染色体上不均匀分布对于scaffolding影响也很大。一个典型的案例就是同源四倍体马铃薯C88的基因组。其经历的杂交、回交历史使得基因组中有大量几乎完全相同的连续区域(可长达超过半条染色体)。这不仅导致scaffolding困难,对于基因组组装来说也是灾难性的。
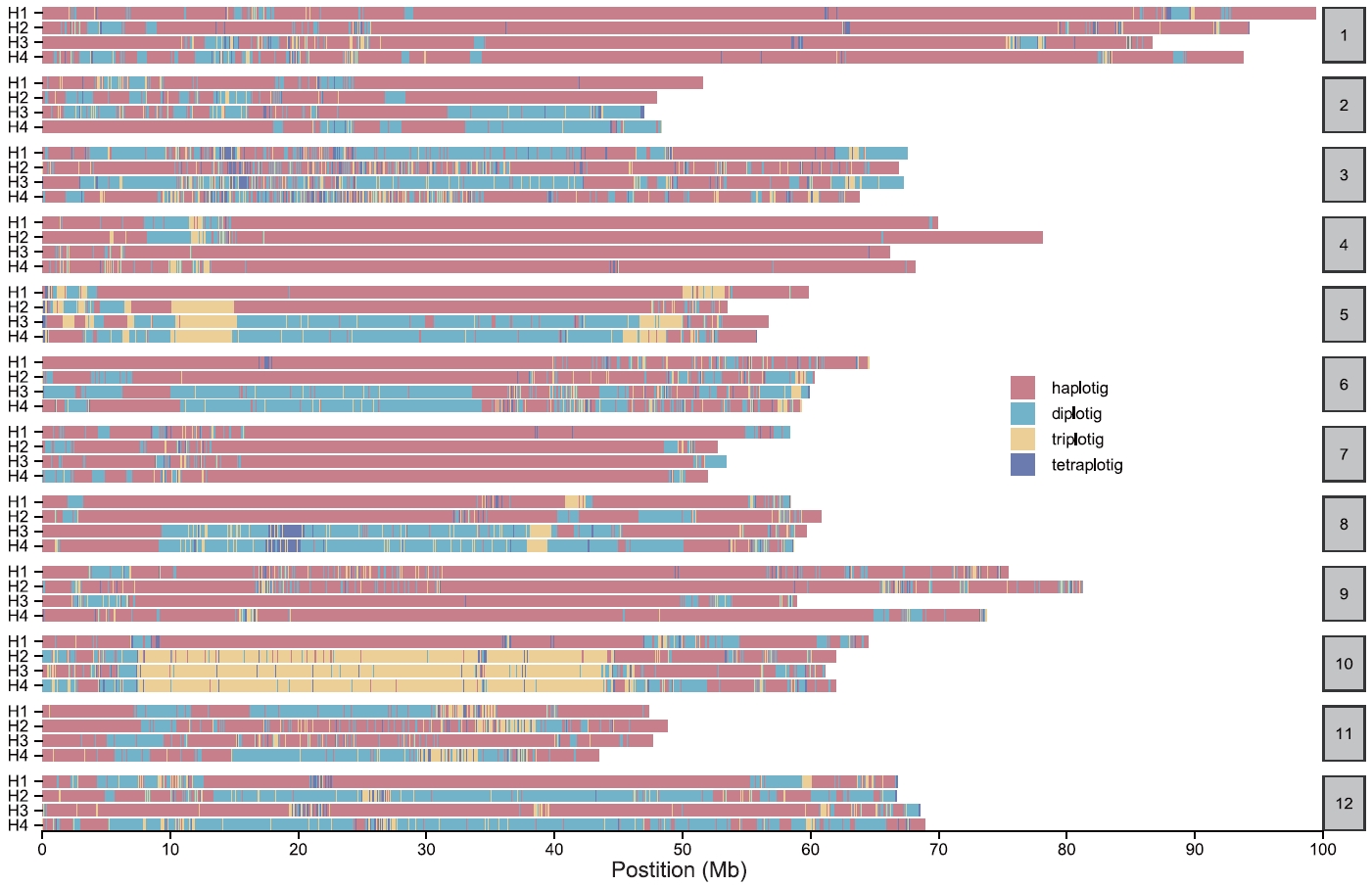
后面这两个难点对于所有的挂载工具都仍是一个巨大的挑战。
基因组大小、Hi-C测序数据量、contig N50、contig数量
HapHiC目前已在超过20 Gb的大基因组中进行了验证。此外,我们的文章中也使用HapHiC尝试挂载了两个大基因组——小麦(14.0 Gb)和银杏(9.9 Gb),并均取得了较好的挂载结果。

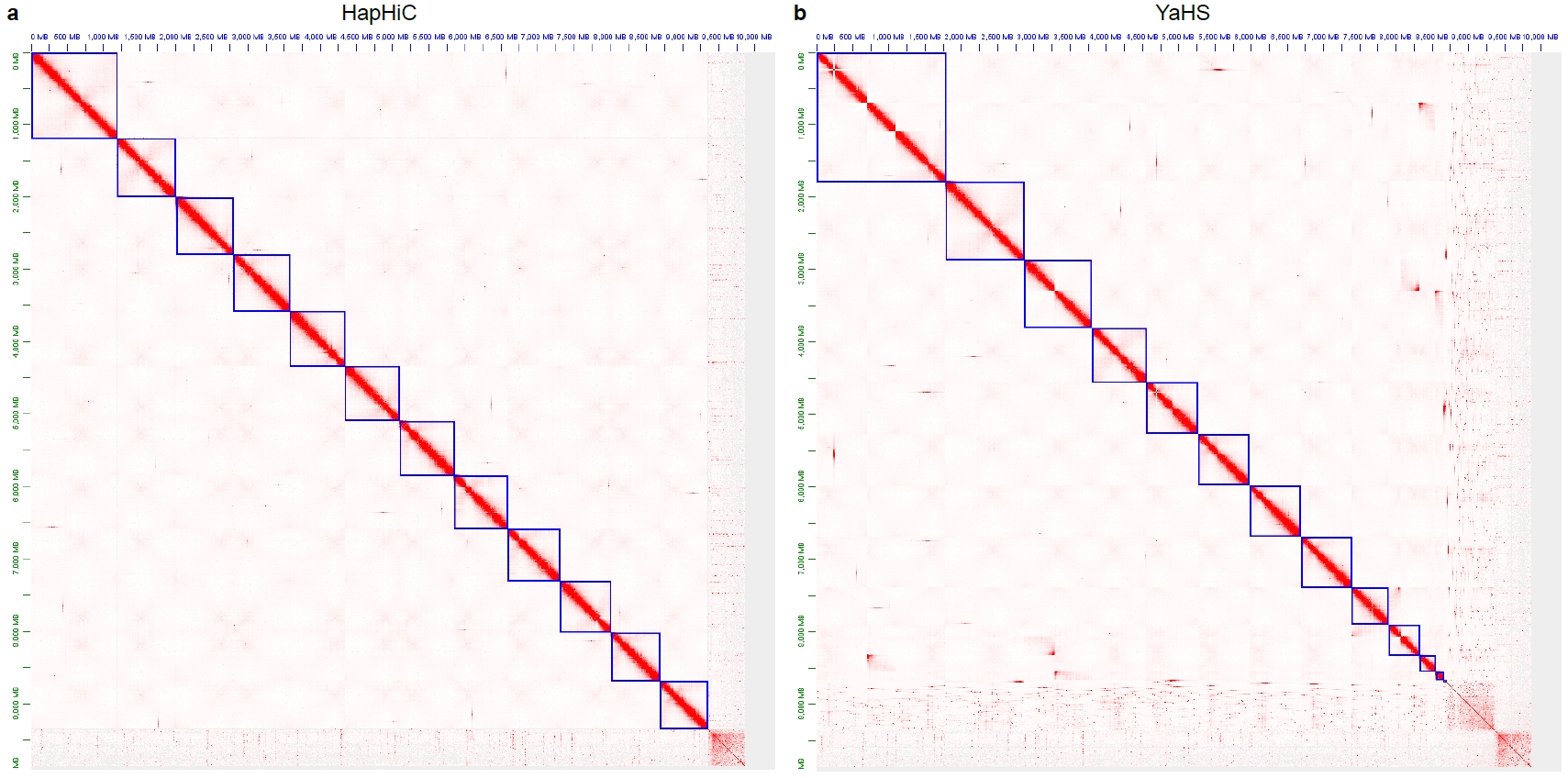
两者分别使用了5.1X和113.5X的Hi-C数据进行挂载:
| T. aestivum (小麦) | G. biloba (银杏) | S. spontaneum AP85-441 (甘蔗) | |
|---|---|---|---|
| 基因组大小(Gb) | 14.0 | 9.9 | 3.1 |
| Contig数量 | 12,982 | 261,820 | 91,977 |
| Contig N50 (bp) | 2,159,703 | 1,579,167 | 44,970 |
| Hi-C数据量(过滤前/过滤后) | 5.1/1.5X | 113.5/54.1X | 93.1/18.5X |
| 实际运行时间(min) | 58.05 | 440.78 | 1243.63 |
| CPU运行时间(min) | 161.25 | 1578.85 | 25851.42 |
| 峰值内存占用(GiB) | 22.98 | 135.83 | 76.38 |
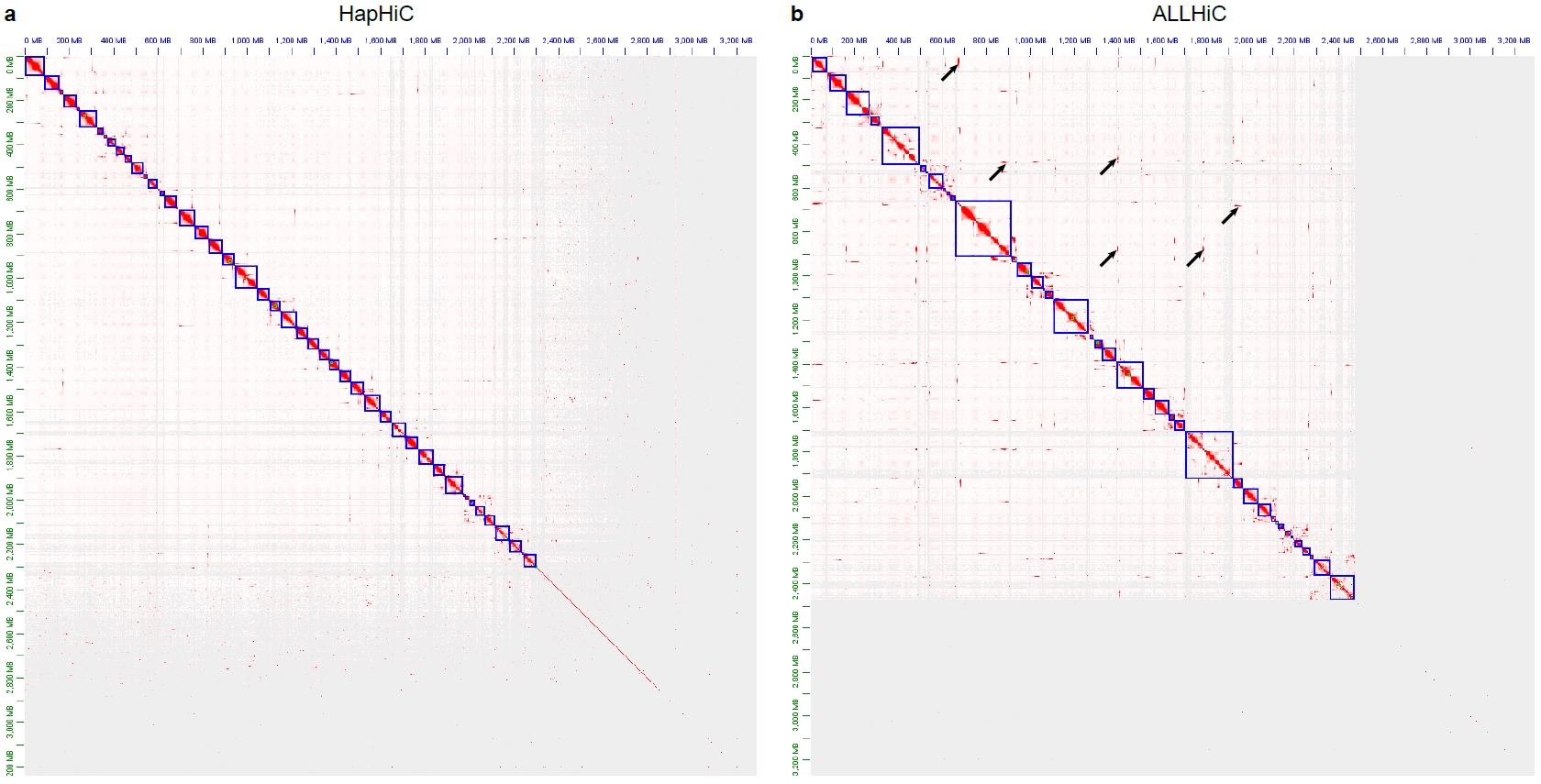
可见,基因组大小并不太影响HapHiC的运行速度和内存占用,主要影响来自Hi-C测序的数据量,以及contig数量。HapHiC针对碎片化的组装结果,在运行速度、内存占用、contig的染色体分配和排序的准确性方面均进行了优化。HapHiC相比仅能处理4,5000条contig的YaHS,可以兼容更多的基因组,提高染色体挂载率。
值得一提的是,在挂载一些小基因组时(比如小于200 Mb),HapHiC的默认参数有可能会无法得到最佳的结果。HapHiC在初步聚类之前会通过默认参数 --Nx 80 过滤长度小于N80的contig片段。但在小基因组中,尤其是contig连续性很高但不同染色体contig长度分布不均匀的时候,这个参数可能会过滤掉完全由短片段组成的染色体。因此建议把这个值设置得更宽松(比如 --Nx 90 ,甚至通过 --Nx 100 完全关闭这个过滤功能)。此外,在第二步——contig的重新分配过程中,HapHiC默认会通过参数 --min_group_len 5 移除小于5 Mb的group,并对其中的contig进行重新分配,以提高连续性和降低额外凝聚层次聚类的错误率。这在大基因组的表现很好,但对于小基因组这个过滤可能太严格,此时建议把这个值设置为 --min_group_len 1 或更低。在后续的更新中,我们可能会用一些自适应的方式使得这两个步骤变得更“聪明”。
返回目录:基因组挂载工具HapHiC:系列教程与方法解读
- 上一篇: 基因组挂载工具HapHiC:系列教程与方法解读
- 下一篇: 没有了
 作者:
作者:
催更
请问作者在对小麦进行挂载时是分步进行的吗?参数都是默认的吗?
用haphic pipeline一步运行的,参数是默认参数,haphic pipeline ${genome} ${bam} 21。
另外最近又参与了另一个小麦的基因组的项目,遇到了排序效果不好的问题,在默认参数基础上增加--skip_allhic参数可解决(只做fast sorting,不做ALLHiC optimization)。
后续没有更新过了么?