肿瘤变异数据分析和可视化工具maftools:突变的数据分析
Maftools系列文章:
上一篇文章《肿瘤变异数据分析和可视化工具maftools:突变数据下载和可视化》主要以TCGA-LUAD为例介绍突变数据和临床数据的下载、处理以及简单的可视化。这篇文章更详细介绍可以利用maftools对肿瘤MAF格式的突变数据做哪些分析。
还和上篇一样,先用maftools把数据读入
具体的数据下载和处理方法这里就不再赘述了,请移步上篇文章:《肿瘤变异数据分析和可视化工具maftools:突变数据下载和可视化》。这篇文章里,读入的临床数据终于可以派上用场了。
library(maftools)
luad <- read.maf(maf="TCGA.LUAD.maf", clinicalData="clinical.tsv")突变的互斥性(exclusive)和共现性(Co-occurrence)分析
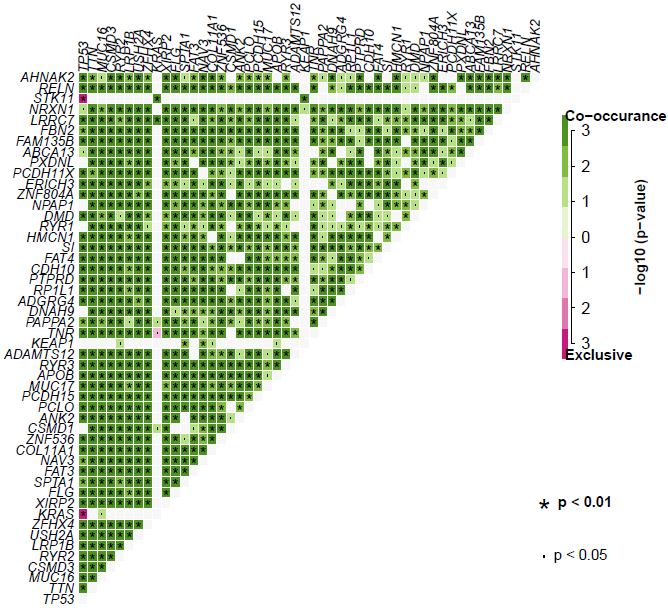
使用函数somaticInteractions可通过对所选突变两两之间进行成对的Fisher精确检验分析突变的互斥性和共现性。此外还通过cometExactTest通过CoMEt检验寻找包含>2基因的互斥基因集。参数top=30用于设定队列中突变最多的30个基因。也可以用参数genes手动设定想要比较的基因列表。pvalue用来设定图中“点”和“星号”标注显著性所对应的阈值。
由于程序原因,直接运行somaticInteractions不能显示成对互斥性和共现性的结果$pairs,需要用一个变量(如下面的output)捕获该函数的返回值并运行两次才能得到$pairs:
> output <- somaticInteractions(maf=luad, top=50, pvalue=c(0.05, 0.01)) # 使用变量捕获函数输出
## Checking for Gene sets
## ------------------
## genes: 4
## geneset size: 3
## 4 combinations
## Signifcantly altered gene-sets: 1
## ------------------
> output # 第一次
## $pairs
##
## $gene_sets
## gene_set pvalue
## 1: TP53, KRAS, STK11 0.04708279
> output # 第二次
## $pairs
## gene1 gene2 pValue oddsRatio 00 11 01 10 Event
## 1: XIRP2 ZFHX4 1.968494e-14 4.850686 334 80 90 61 Co_Occurance
## 2: MUC16 TTN 3.880676e-14 3.817014 229 146 112 78 Co_Occurance
## 3: LRP1B MUC16 4.388807e-14 4.074352 272 114 110 69 Co_Occurance
## 4: RYR2 LRP1B 6.666331e-14 4.068623 284 107 76 98 Co_Occurance
## 5: TTN RYR2 9.467845e-14 3.835079 238 136 69 122 Co_Occurance
## ---
## 1039: AHNAK2 RP1L1 4.506738e-02 1.853890 415 19 72 59 Co_Occurance
## 1040: ZNF536 SPTA1 4.702016e-02 1.613526 353 35 102 75 Co_Occurance
## 1041: LRRC7 FAT4 4.792946e-02 1.807431 414 19 69 63 Co_Occurance
## 1042: ADGRG4 ADAMTS12 4.907451e-02 1.769172 398 23 76 68 Co_Occurance
## 1043: RELN ADGRG4 4.978755e-02 1.784468 413 19 72 61 Co_Occurance
##
## $gene_sets
## gene_set pvalue
## 1: TP53, KRAS, STK11 0.04708279本例中共获得1042个显著互斥/共现的突变基因对(Fisher精确检验)以及1个互斥的基因集(CoMEt检验)。注意程序输出的“occurance”存在拼写错误,应为“occurrence”。输出详细结果可以使用write.table写入到文件:
write.table(output$pairs, file="somaticInteractions.pairwise.tsv", quote=FALSE, row.names=FALSE, sep="\t")
write.table(output$gene_sets, file="somaticInteractions.comet.tsv", quote=FALSE, row.names=FALSE, sep="\t")生成的图以矩阵的形式展示基因对Fisher精确检验的结果。其中绿色和洋红色分别代表共现和互斥,颜色深浅代表显著性程度-log10(p-value):
然后可以选择感兴趣的基因,使用oncostrip展示(当然也可以使用oncoplot):
oncostrip(maf=luad, genes=c("TP53", "KRAS", "STK11"), border=NULL) # 这里选择了exclusive的基因集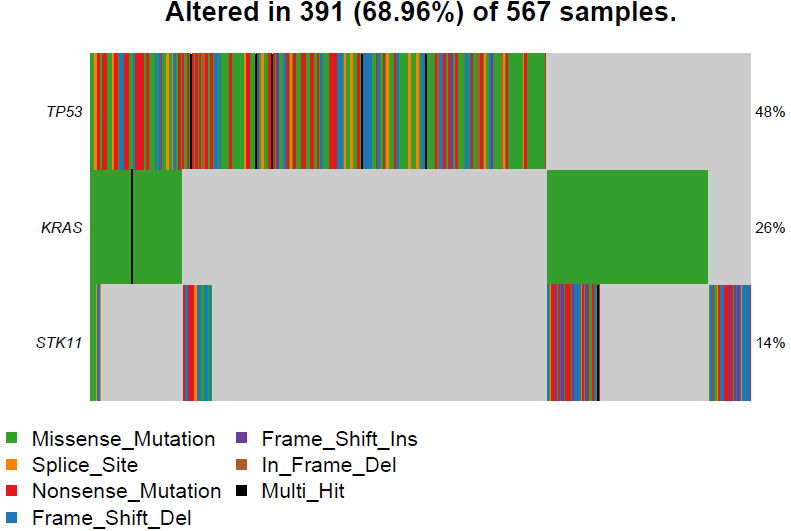
预测癌症驱动基因
maftools中的函数oncodrive基于“oncodriveCLUST算法”可直接从MAF文件中鉴定癌症驱动基因。其原理是癌症驱动基因的突变通常富集在特定位点(热点):
luad.sig <- oncodrive(maf=luad, minMut=5, AACol="HGVSp_Short", pvalMethod="zscore")
# 将统计结果保存到文件
write.table(luad.sig, file="luad.oncodrive.tsv", quote=FALSE, row.names=FALSE, sep="\t")
# 使用plotOncodrive函数绘制散点图
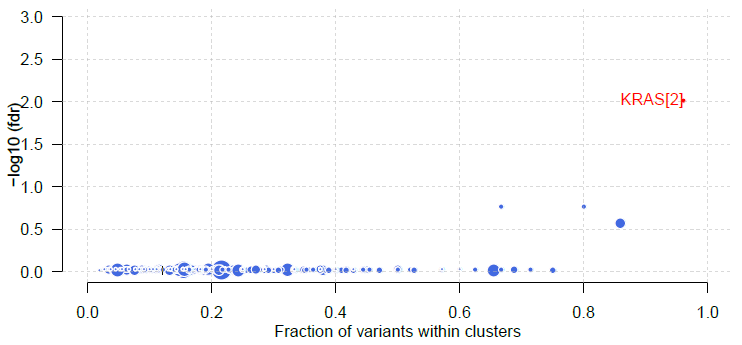
plotOncodrive(res = laml.sig, fdrCutOff = 0.1, useFraction = TRUE)图中点的大小以及基因名后的数字展示的是基因内突变cluster的数量,X轴为cluster内的突变数量或者占突变总数的比例(根据参数useFraction设定显示方式),Y轴是-log10(fdr)。本例(TCGA-LUAD)中鉴定到的驱动基因candidate为KRAS:
pfam注释和统计
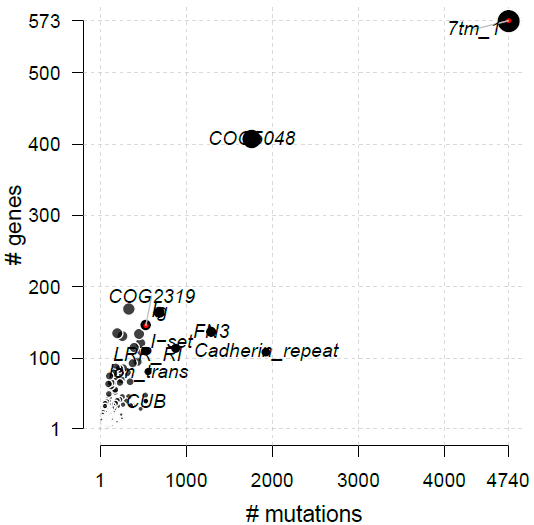
pfamDomains可以用来注释导致氨基酸改变的突变所在pfam结构域以及按照pfam结构域为对象的统计结果(分别储存在luad.pfam$proteinSummary和luad.pfam$domainSummary中),并绘制散点图:
luad.pfam <- pfamDomains(maf=luad, AACol="HGVSp_Short", top=10)圆点代表pfam结构域,对应X轴为该结构域中出现的突变数量,Y轴以及圆点的大小为影响的基因数。参数top用来选择标记突变数最多的pfam domain数量。
统计结果的展示和保存:
> luad.pfam$proteinSummary[,1:7, with=FALSE]
## HGNC AAPos Variant_Classification N total fraction DomainLabel
## 1: KRAS 12 Missense_Mutation 136 154 0.88311688 COG1100
## 2: EGFR 858 Missense_Mutation 23 78 0.29487179 PTKc_EGFR
## 3: EGFR 746 In_Frame_Del 17 78 0.21794872 PTKc_EGFR
## 4: TP53 273 Missense_Mutation 10 281 0.03558719 P53
## 5: TP53 249 Missense_Mutation 10 281 0.03558719 P53
## ---
## 138224: pk 753 Missense_Mutation 1 6 0.16666667 <NA>
## 138225: pk 543 Missense_Mutation 1 6 0.16666667 <NA>
## 138226: pk 735 Missense_Mutation 1 6 0.16666667 <NA>
## 138227: pk 316 Missense_Mutation 1 6 0.16666667 <NA>
## 138228: pk 313 Missense_Mutation 1 6 0.16666667 <NA>
> luad.pfam$domainSummary[,1:3, with=FALSE]
## DomainLabel nMuts nGenes
## 1: 7tm_1 4740 573
## 2: Cadherin_repeat 1925 109
## 3: COG5048 1760 408
## 4: FN3 1290 138
## 5: I-set 878 115
## ---
## 6734: zf-Sec23_Sec24 1 1
## 6735: zf-ZPR1 1 1
## 6736: zf-piccolo 1 1
## 6737: zf-primase 1 1
## 6738: zf-rbx1 1 1
> write.table(luad.pfam$proteinSummary, file="pfam_protsum.tsv", quote=FALSE, row.names=FALSE, sep="\t")
> write.table(luad.pfam$domainSummary, file="pfam_domainsum.tsv", quote=FALSE, row.names=FALSE, sep="\t")泛癌的比较分析
pancanComparison函数可以将给定的队列和21个癌症队列中高频突变的基因(参考文献)进行比较,从而分析给定队列中特异性的突变。做这个分析需要用到MutSigCV的结果。(略,有时间再补充)
生存分析
2020/03/03更新:
- 生存分析这块,maftools的作者可能在几个月前修改过代码,输入参数发生了变化,因此这一部分本问的内容会和最新版不太一样,如果你安装的github的最新版maftools,请以maftools的官方文档操作为准。
- 参考GitHub issue:https://github.com/PoisonAlien/maftools/issues/409
使用函数mafSurvival可以利用突变和临床数据绘制Kaplan-Meier曲线(KM曲线)进行生存分析。临床数据的处理请翻看上篇文章《肿瘤变异数据分析和可视化工具maftools:突变数据下载和可视化》。参数genes用于选定基因,time设定临床数据中保存随访时间的字段,Status设定临床数据中存放生存状态的。
参数isTCGA在这里设定数据是否来自TCGA。如果设定为TRUE,程序会将临床数据中"Tumor_Sample_Barcode"字段前12个字符截取出来和MAF文件进行匹配。因为我在上篇文章中通过一个Python脚本tcga_format.py已经做过这个处理了,所以这里是否设定TRUE都可以(未免麻烦,建议这么做)。
mafSurvival(maf=luad, genes=c("TP53", "TTN", "MUC16"), time="days_to_last_follow_up", Status="days_to_death", isTCGA=TRUE)输出突变/野生型样本数量、生存时间中位数等信息:
## Looking for clinical data in annoatation slot of MAF..
## Number of mutated samples for given genes:
## TP53 TTN MUC16
## 270 258 224
## Median survival..
## Group medianTime N
## 1: Mutant 606 414
## 2: WT 704 153
## Removed 178 samples with NA'sKM曲线和logrank test:
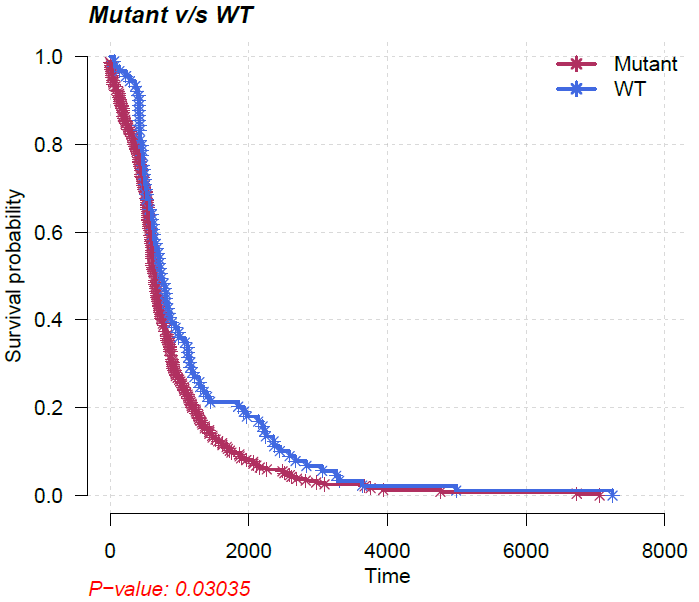
比较两个MAF文件(队列)
函数mafCompare通过对两个队列的MAF文件中所有基因进行Fisher精确检验检测差异突变基因。Maftools官方文档给了一个例子是Acute Promyelocytic Leukemia(APL,急性早幼粒细胞白血病)在不同分期两个队列中的差异突变基因。2016年发表在Cancer Cell上的一篇文章中分析了癌症中的性别差异。其中LUAD也是性别偏好性明显的癌症类别。这里我拿不同性别的TCGA-LUAD进行比较分析:
# 从临床数据中提取性别对应的"Tumor_Sample_Barcode"
clin <- read.table("clinical.tsv", header=T, sep="\t")
clin.female <- subset(clin, gender=="female")$Tumor_Sample_Barcode
clin.male <- subset(clin, gender=="male")$Tumor_Sample_Barcode
# 使用subsetMaf构建男性和女性的MAF对象
luad.female <- subsetMaf(maf=luad, tsb=clin.female, isTCGA=TRUE)
# 使用mafCompare比较差异突变基因
fvsm <- mafCompare(m1=luad.female, m2=luad.male, m1Name="Female", m2Name="Male", minMut=5)
# 结果保存到文件"female_vs_male.tsv"
write.table(fvsm$results, file="female_vs_male.tsv", quote=FALSE, row.names=FALSE, sep="\t")1. 绘制森林图
续上一个例子,用forestPlot可以绘制森林图展示分析结果:
forestPlot(mafCompareRes=fvsm, pVal=0.001, color=c("maroon", "royalblue"), geneFontSize=0.8)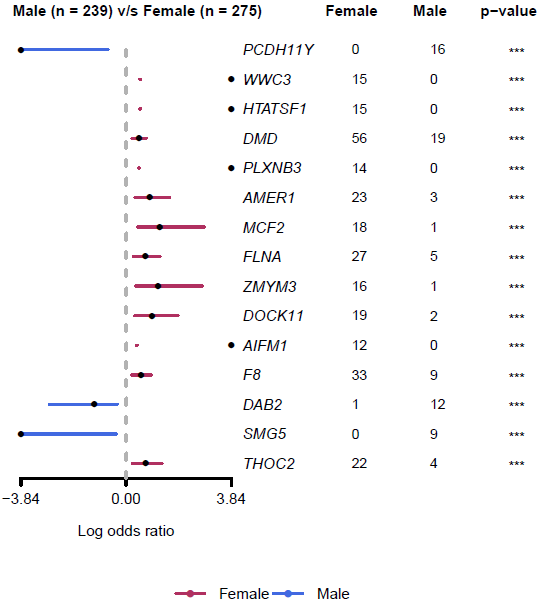
2. 比较两个队列的oncoplot
使用coOncoplot并排绘制两个队列的oncoplot:
genes <- c("PCDH11Y", "HTATSF1", "WWC3", "DMD", "PLXNB3", "AMER", "MCF2", "FLNA")
coOncoplot(m1=luad.female, m2=luad.male, m1Name="Female", m2Name="male", genes=genes)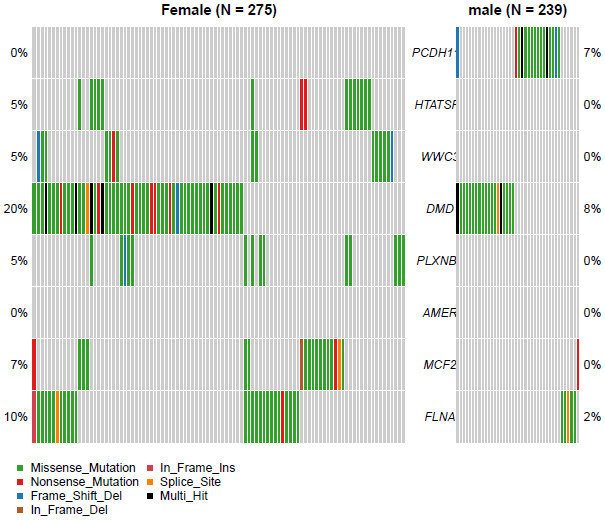
3. 比较两个队列的棒棒糖图
使用lollipopPlot2比较两个队列突变位点和类型:
lollipopPlot2(m1=luad.female, m2=luad.male, m1_name="Female", m2_name="Male", gene="IQGAP2", AACol1 = "HGVSp_Short", AACol2 = "HGVSp_Short")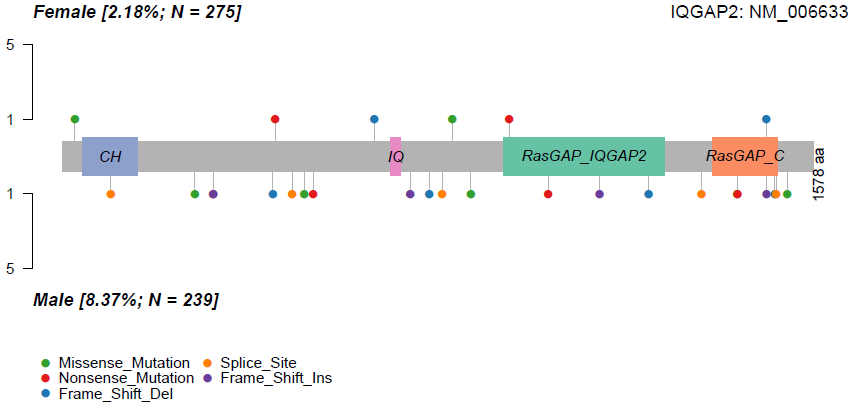
临床富集分析
clinicalEnrichment可用于做临床数据的富集分析(突变富集在哪些临床特征上),和上面例子类似,这里对性别进行富集:
clin_enrich <- clinicalEnrichment(maf=luad, clinicalFeature="gender")clinicalEnrichment会进行两两配对(比如female vs male)和分组的Fisher精确检验(female vs rest),结果分别储存于$pairwise_comparision和$groupwise_comparision之中。这里就不贴返回结果了,用writetable保存到文件:
write.table(clin_enrich$pairwise_comparision, file="clin_enrich_pair.tsv", quote=FALSE, row.names=FALSE, sep="\t")
write.table(clin_enrich$groupwise_comparision, file="clin_enrich_group.tsv", quote=FALSE, row.names=FALSE, sep="\t")绘制富集结果,pVal用来控制输出pvalue的阈值,似乎目前还不能按照fdr进行筛选:
plotEnrichmentResults(enrich_res=clin_enrich, pVal=0.001)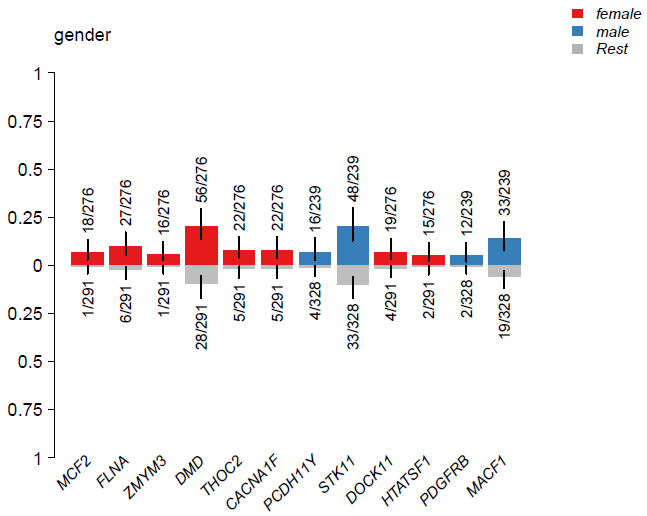
这里的分析做得比较粗糙,因为没有去掉未报道性别的case,建议先对临床特征进行筛选再使用,否则会影响groupwise的结果。
药物基因互作
drugInteractions函数可以通过查询编译到maftools中的Drug Gene Interaction database分析基因的成药性(gene druggability)以及药物和基因间的互作。注意:本函数用到的数据来源为纯科研用途,不构成医疗建议。
1. 基因的成药性
druggability <- drugInteractions(maf=luad)
# 保存到文件
write.table(druggability, file="druggability.tsv", quote=FALSE, row.names=FALSE, sep="\t")
图中展示潜在的可成药基因分类,每个分类后面中括号里的是前5的基因,少于5个就全部显示。Y轴为可成药基因分类中的基因数量。
2. 药物和基因间互作
通过给定基因或基因列表,查询相应互作的药物:
kras <- drugInteractions(genes="KRAS", drugs=TRUE)
# 展示其中部分列
kras[,.(Gene, interaction_types, drug_name, drug_claim_name)]
# 保存到文件
write.table(kras, file="kras_drug.tsv", quote=FALSE, row.names=FALSE, sep="\t")输出结果:
## Gene interaction_types drug_name drug_claim_name
## 1: KRAS LENALIDOMIDE lenalidomide
## 2: KRAS IMGATUZUMAB GA201
## 3: KRAS 3144
## 4: KRAS SELUMETINIB Selumetinib
## 5: KRAS BUPARLISIB BKM120
## ---
## 291: KRAS GEDATOLISIB Gedatolisib
## 292: KRAS XMT-1536
## 293: KRAS GEMCITABINE Gemcitabine
## 294: KRAS NAVITOCLAX ABT-263
## 295: KRAS PA-799 CH5132799致癌信号通路
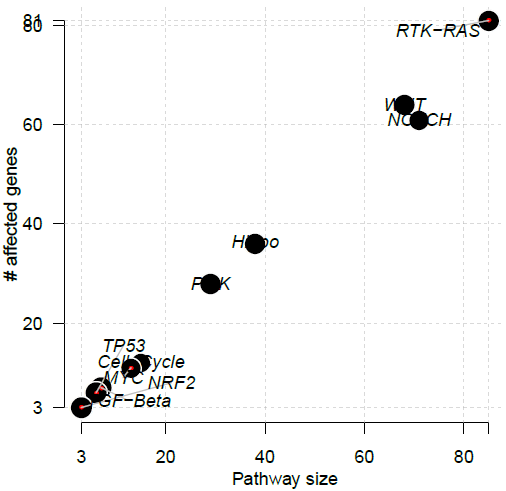
OncogenicPathways函数可以分析TCGA中已知的10个致癌信号通路中基因突变的数量和比例。包括cell cycle, Hippo, Myc, Notch, Nrf2, PI-3-Kinase/Akt, RTK-RAS, TGFβ signaling, p53 and β-catenin/Wnt:
OncogenicPathways(maf=luad)输出致癌信号通路基因突变统计:
## Pathway alteration fractions
## Pathway N n_affected_genes fraction_affected
## 1: RTK-RAS 85 81 0.9529412
## 2: WNT 68 64 0.9411765
## 3: NOTCH 71 61 0.8591549
## 4: Hippo 38 36 0.9473684
## 5: PI3K 29 28 0.9655172
## 6: Cell_Cycle 15 12 0.8000000
## 7: MYC 13 11 0.8461538
## 8: TGF-Beta 7 7 1.0000000
## 9: TP53 6 6 1.0000000
## 10: NRF2 3 3 1.0000000绘制的散点图:
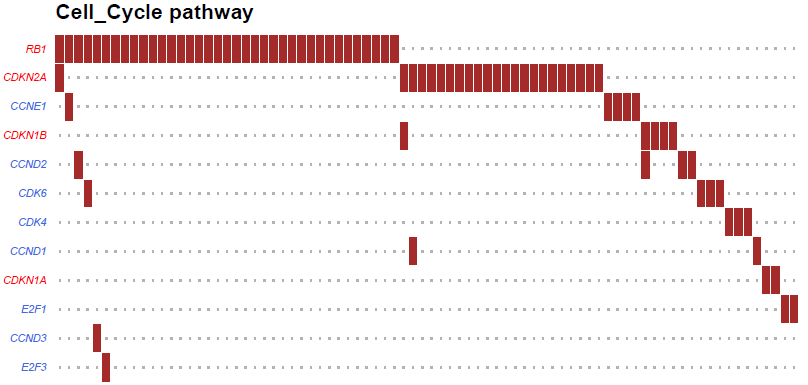
PlotOncogenicPathways函数也可以用瀑布图的形式展示某个pathway所有基因的图片情况,其中红色标注的基因为抑癌基因,蓝色标注的为致癌基因:
肿瘤异质性和MATH score
1. 肿瘤样品的异质性
肿瘤通常是异质性的,包含多种克隆。通过对等位基因频率VAF的聚类可以推断肿瘤的异质性。函数inferHeterogeneity可以实现这个功能(需要调用mclust):
# 指定Tumor_Sample_Barcodes为"TCGA-55-7283"
vafclust <- inferHeterogeneity(maf=luad, tsb="TCGA-55-7283")
# 保存结果到文件
write.table(vafclust$clusterData, file="vaf_clustdata.tsv", quote=FALSE, row.names=FALSE, sep="\t")
write.table(vafclust$clusterMeans, file="vaf_clustmean.tsv", quote=FALSE, row.names=FALSE, sep="\t")输出结果中各突变基因VAF的数据储存在$clusterData中,聚类数量和平均VAF储存在$clusterMeans中。用plotClusters函数展示聚类结果:
plotClusters(clusters=vafclust)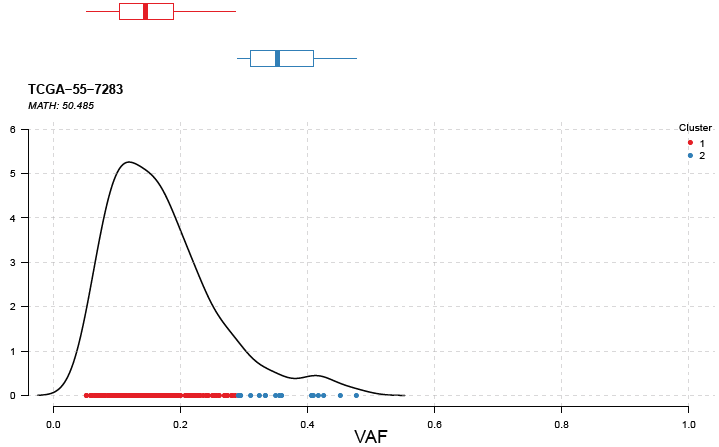
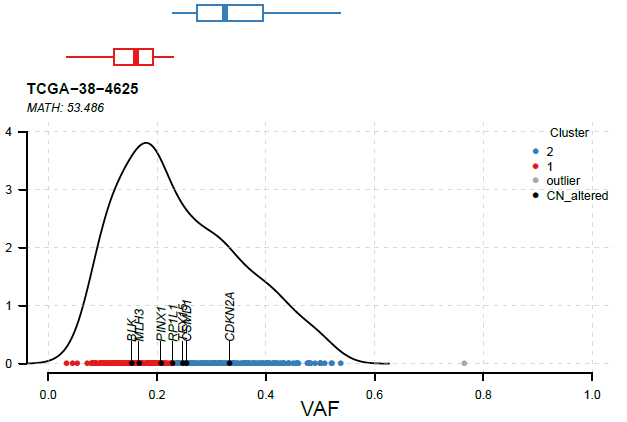
可以看到聚成两类,说明可能存在两种克隆。图上方的箱线图展示聚类中的数据分布。除了聚类的方法,还有一种用来量化肿瘤异质性的方式叫做"MATH score",用来计算VAF分布的宽度(即离散程度)。通常很高的MATH score与更差的预后相关。
2. 排除发生了拷贝数变异的突变基因
因为突变基因的CNV会影响到VAF的计算,因此在做聚类之前最好最好排除这部分基因。我们需要在TCGA上下载样品对应的DNACopy生成的Copy Number Segment文件。并把"GDC_Aliquot"字段替换为"Sample",该列内容也替换为对应的Tumor_Sample_Barcodes。这里以TCGA-38-4625为例:
sed 's/GDC_Aliquot/Sample/g' LOOBY_p_TCGAb58_SNP_N_GenomeWideSNP_6_E04_680252.nocnv_grch38.seg.v2.txt | sed 's/d0597d1
4-df0c-413b-888f-53597d1fb61a/TCGA-38-4625/g' > TCGA-38-4625.seg重新运行inferHeterogeneity和plotClusters
vafclust <- inferHeterogeneity(maf=luad, tsb="TCGA-38-4625", segFile="TCGA-38-4625.seg")
plotClusters(clusters=vafclust, genes='CN_altered', showCNvars=TRUE)inferHeterogeneity将根据读入的Segment文件去掉没有CNV数据的突变并标记存在CNV的突变。
突变特征
1. 构建突变矩阵 & 计算APOBEC enrichment score
分析突变特征需要读入突变位点上下游的序列构建突变矩阵,因此需要使用到全基因组序列。这里因为作为案例的TCGA-LUAD使用的参考基因组是hg38,因此使用"BSgenome.Hsapiens.UCSC.hg38",如果参考基因组为hg19,则用对应的"BSgenome.Hsapiens.UCSC.hg19":
BiocManager::install("BSgenome.Hsapiens.UCSC.hg38")
library(BSgenome.Hsapiens.UCSC.hg38, quietly=TRUE)
# 预测APOBEC enrichment scores & 构建用于突变特征分析的突变矩阵
luad.tnm <- trinucleotideMatrix(maf=luad, ref_genome="BSgenome.Hsapiens.UCSC.hg38")2. APOBEC富集和非富集样品差异
apobec_enrich <- plotApobecDiff(tnm=luad.tnm, maf=luad)
# 保存到文件
write.table(apobec_enrich$results, file="apobec_result.tsv", quote=FALSE, row.names=FALSE, sep="\t")
write.table(apobec_enrich$SampleSummary, file="apobec_sum.tsv", quote=FALSE, row.names=FALSE, sep="\t")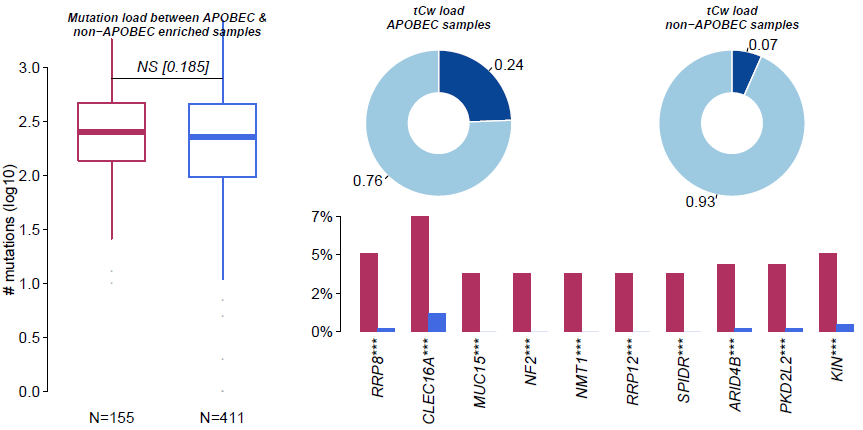
3. 突变特征分析
extractSignatures函数用于从96种替换类型中提取突变特征。这里nTry=6设定尝试n的最大值,程序将调用NMF计算cophenetic correlation coefficient并选取最合适的值。因为内存消耗太大,后面的例子都没跑通,有时间再补上:
library("NMF")
luad.sign <- extractSignatures(mat=luad.tnm, nTry=6, plotBestFitRes=FALSE)
plotSignatures(luad.sign)绘制成热图:
library('pheatmap')
pheatmap::pheatmap(mat=luad.sign$coSineSimMat, cluster_rows=FALSE, main="cosine similarity against validated signatures")4. 突变特征富集分析
luad.se <- signatureEnrichment(maf=luad, sig_res=luad.sign)plotEnrichmentResults(enrich_res=luad.se, pVal=0.05)参考资料
https://www.bioconductor.org/packages/release/bioc/vignettes/maftools/inst/doc/maftools.html
 作者:
作者:
你好,MATH代表异质性我用到GBM上效果不好,高低组生存没影响,而且IDH WT(提示不良预后)相比IDH mut的MATH更低,是不是MATH这个算法不是很理想?谢谢
我认为肿瘤异质性和预后不一定相关,也不能将得不到预期的结果称作“不理想”
你好,请问使用subsetMaf后 luad@clinical.data$Tumor_Sample_Barcode 中变量个数为0,该怎么解决呢?
check一下你前面各步骤是不是对的吧,把变量打印出来看看
你好,我按照步骤同样也是下载了LUAD的maf和临床数据,但无法使用mafSurvival来进行生存分析,请问应该怎么处理下载下来的临床资料呀。我按照教程将submitter_id改成了Tumor_Sample_Barcode了,但还是不行。显示“Empty data.table(0 rows and 3cols):Group, medianTime,N”。所以我想应该是maf文件和临床资料没有能融合一起。想向大神请教一下
大神你好,在绘制森林图的时候发现报错信息,不知道指的时哪里的问题呀?
fvsm <- mafCompare(m1=luad.female, m2=luad.male, m1Name="female", m2Name="male", minMut=5)
Error in (function (classes, fdef, mtable) :
unable to find an inherited method for function ‘getGeneSummary’ for signature ‘"data.table"’
大神不敢当,检查你luad.female和luad.male变量的值和变量类型吧
想问问博主,在后来 mutation signature数据跑通了吗,我在跑的时候extractSignatures()的时候,用普通自己数据可以,不知道为什么跑TCGA数据的时候 ,报错 Error in (new("standardGeneric", .Data = function (x) :
参数没有用(model = list("NMFstd", 3, 0), method = "random"), 想请教一下什么问题,我用hg38作为reference和lifover后用hg19做reference都这样呢。。
最近手头的电脑应该配置够了,本周末可能会更新一下
后续我又重新尝试了一下,hg38参考基因集只要library之后,就跑不通了,可能是某些函数赋值冲突造成的,建议都使用hg19的注释格式,并且detach UCSC.hg38参考基因集.
感谢你的反馈!最近实在事情太多,没有来得及更新
前辈,我按照处理表格的方法把临床资料处理了,但是到最后重新尝试分析生存资料使用mafSurvival命令的时候,不知道为什么出错,提示了一下信息:
Median survival..
Group medianTime N
1: WT 626 772
Error in survdiff.fit(y, groups, strata.keep, rho) :
There is only 1 group
就是说没有了Mutant那个组,我这里想不明白,按道理不可能没有了Mutant那个组的呀。
请教前辈
关于你生存分析的几个问题,这里统一回复一下:maftools的作者可能在几个月前修改过代码,你提到用0/1代替alive/dead是近期修改的内容,你的操作方式应该也是follow了新版的文档,但是可能你的maftools包是旧的,建议你安装github的最新版。参考:https://github.com/PoisonAlien/maftools/issues/409
非常感谢分享!请问计算MATH的seg文件是每个Tumor_Sample_Barcodes对应一个文件吗?那很难批量获得吧
非常感谢您的分享,我也是做肿瘤基因组数据分析的,可以留个邮箱交流问题吗