肿瘤变异数据分析和可视化工具maftools:突变数据下载和可视化
Maftools系列文章:
本文接上次的内容:《肿瘤变异数据分析和可视化工具maftools:安装和文件格式要求》,本文以TCGA肺腺癌(LUAD)的数据为例介绍突变数据下载和可视化。
数据下载和处理
在TCGA官网下载TCGA-LUAD项目中mutect2输出的MAF格式的突变以及临床信息:
$ ls -lhrt
total 50M
-rw-r--r-- 1 xiaofei xiaofei 50M May 14 22:44 TCGA.LUAD.maf.gz
-rw-r--r-- 1 xiaofei xiaofei 157K May 15 00:12 clinical.tsv因为要通过"Tumor_Sample_Barcode"列将两个文件进行关联,这里做两步处理:
- 将临床信息文件
clinical.tsv中存放样品ID的"submitter_id"修改为"Tumor_Sample_Barcode"。 - 把
TCGA.LUAD.maf.gz中的"Tumor_Sample_Barcode"列按照clinical.tsv中的修改成一致。具体在此例中就是只保留"Project-TSS-Participant"部分。这里我使用自己写的Python脚本进行修改(代码获取:GitHub):
$ python tcga_format.py TCGA.LUAD.maf.gz > TCGA.LUAD.maf注:虽然在本篇文章中还没用到临床信息,不过姑且先了解一下数据读入前应该如何处理。
使用maftools读取MAF文件并统计
1. MAF文件读取
library(maftools)
luad <- read.maf(maf="TCGA.LUAD.maf", clinicalData="clinical.tsv")文件读入时的输出(不同版本的maftools输出结果会有所不同):
## -Reading
## |--------------------------------------------------|
## |==================================================|
## |--------------------------------------------------|
## |==================================================|
## -Validating
## -Silent variants: 66099
## -Summarizing
## --Possible FLAGS among top ten genes:
## TTN
## MUC16
## USH2A
## -Processing clinical data
## -Finished in 15.2s elapsed (16.3s cpu) 2. MAF文件统计
使用如下代码可以对读入的MAF文件进行汇总统计:
luad # 直接输入MAF对象可以查看MAF文件的基本信息
getSampleSummary(luad) # 显示样品的统计
getGeneSummary(luad) # 显示基因的统计
getClinicalData(luad) # 显示样品关联的临床数据
getFields(luad) # 显示MAF文件中的所有字段
write.mafSummary(maf=luad, basename="luad") # 将详细统计结果输出到文件具体输出结果这里就不展示了,可以自行尝试。
突变的可视化
1. MAF文件汇总统计图
使用如下代码即可绘制MAF文件的汇总统计图。dashboard style中包括"Variant Classification"、"Variant Type"、"SNV Class"、"Variants per sample"、"Variant Classification summary"、"Top 10 mutated genes"的统计:
plotmafSummary(maf=luad, rmOutlier=TRUE, addStat="median", dashboard=TRUE, titvRaw = FALSE)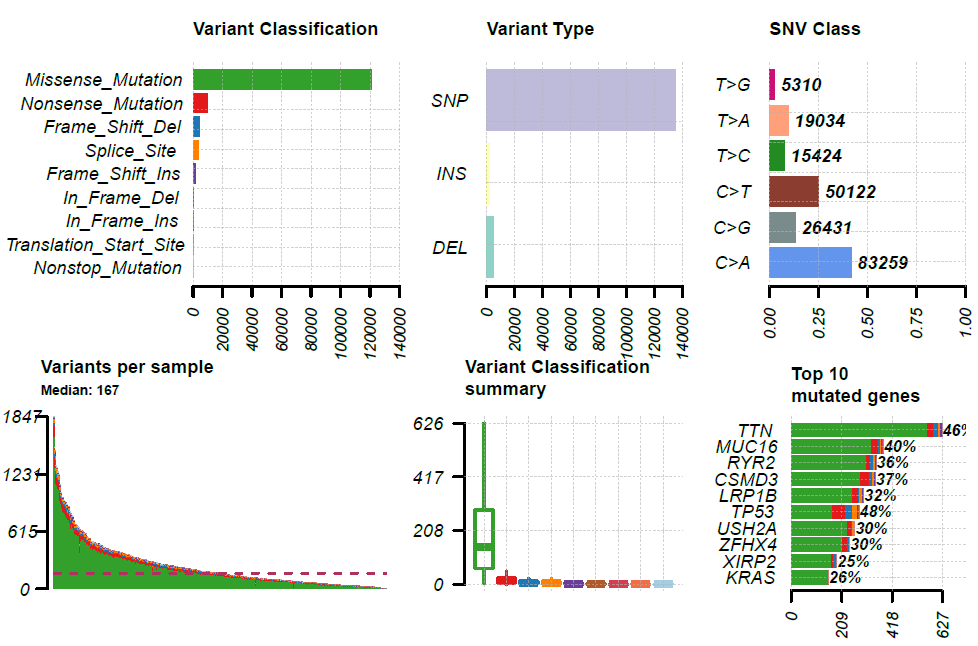
2. Oncoplots
Oncoplot也就是cBioPortal中的OncoPrint或者叫做瀑布图。简单使用方法如下,通过top=10设定要展示的频率前10的突变:
oncoplot(maf=luad, top=10, borderCol=NULL)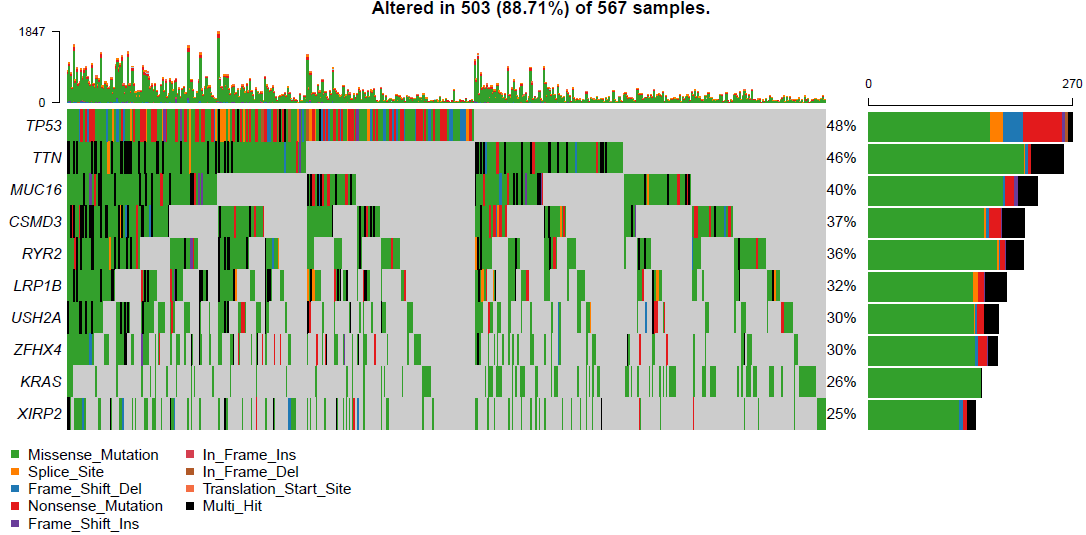
因为sample数比较多,图缩小之后不是很好看,甚至显示错误。这里可以设置参数borderCol=NULL让样本之间不显示边界,推荐大样本量的时候使用(cBioPortal显示类似,会自适应进行调整)。
此外,Oncoplot的定制化可通过调整参数和输入的MAF对象实现:
- 通过参数
genes只展示某几个感兴趣的基因的突变情况 draw_titv=TRUE: 在oncoplot中增加转换和颠换的统计colors: 更改配色- 如果读入MAF文件时也输入了GISTIC的CNV文件,或者有其他格式的CNV文件,可以在oncoplot中展示
- 可以在oncoplot中加入显著性概率(比如MutSig的结果)、表达量、或者任何其他数值
- 可以通过
clinicalFeatures参数加入临床数据的注释/分类信息并以sortByAnnotation进行排序 - 更多方法详见:http://bioconductor.org/packages/devel/bioc/vignettes/maftools/inst/doc/oncoplots.html
3. Oncostrip
oncostrip本质上和oncoplot没什么区别,代码实现也就是oncoplot的封装,只是drawRowBar和drawColBar设置成了FALSE。这里就不做展示了。
4. 转换(transition)和颠换(transversion)的统计和可视化
使用titv函数对SNP进行转换和颠换的分类,然后使用plotTiTv进行可视化:
luad.titv <- titv(maf=luad, plot=FALSE, useSyn=TRUE)
plotTiTv(res=luad.titv)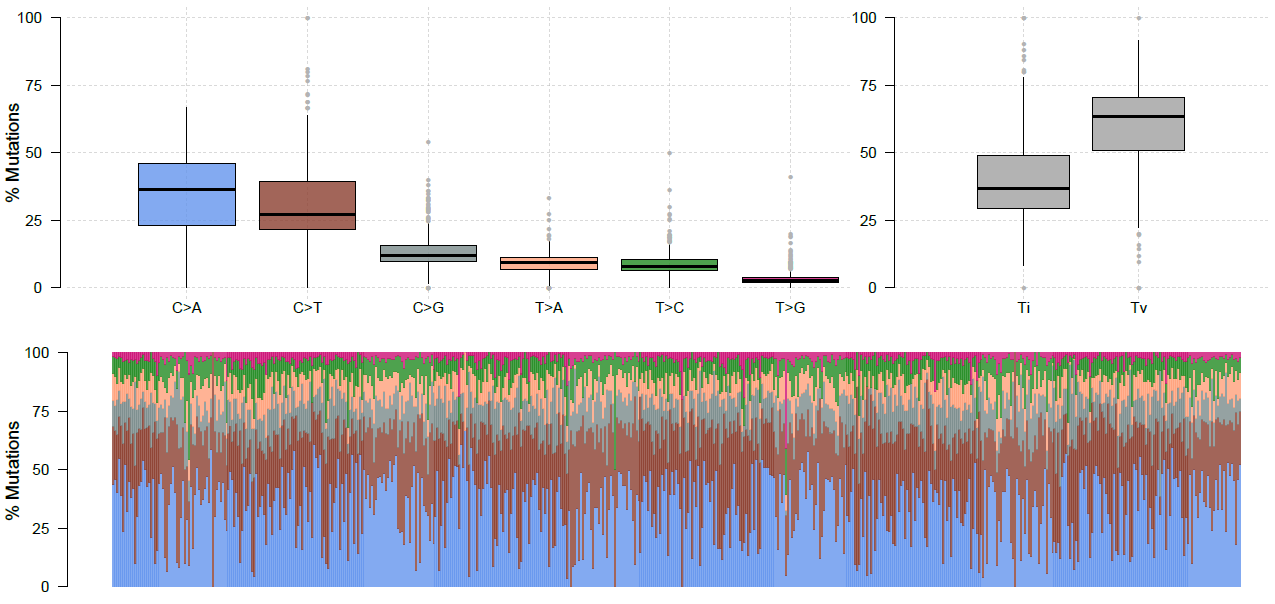
5. Lollipop图(棒棒糖图)
使用lollipopPlot函数可以绘制棒棒糖图,用于显示蛋白的结构域以及氨基酸突变的位点和类型。这个功能需要MAF文件中有体现氨基酸变化的字段,并通过AACol参数进行设定(这个字段通常没有固定的名称)。本例中手动指定该字段为HGVSp_Short:
lollipopPlot(maf=luad, gene="TP53", AACol="HGVSp_Short", showMutationRate=TRUE)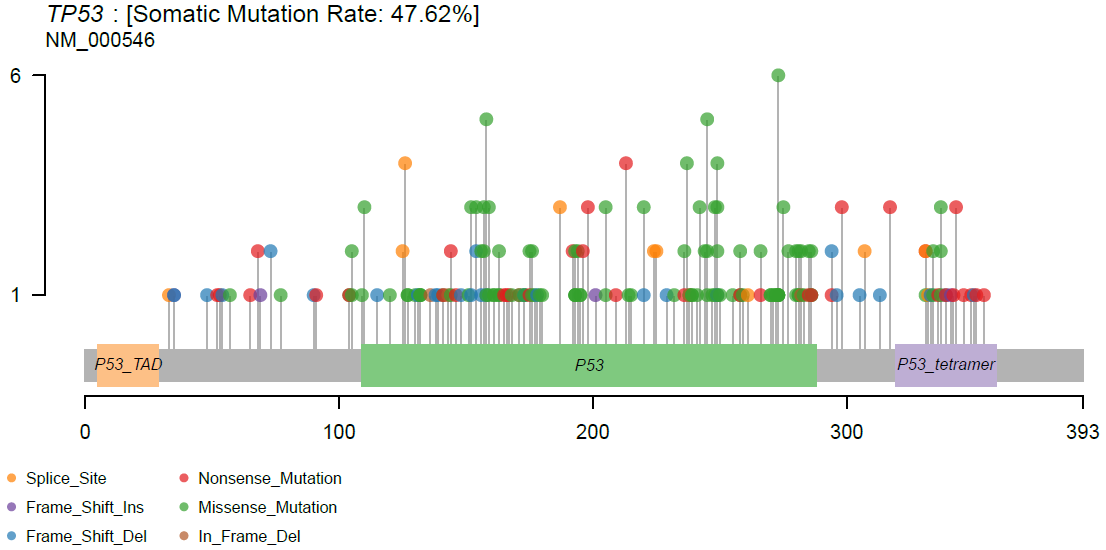
此外可以使用labelPos参数标记突变。
6. Rainfall plots
rainfallPlot可以通过绘制染色体尺度的突变间距的展示hyper mutated genomic region。因为使用TCGA-LUAD的数据没有找到"Kataegis",这里用程序自带的乳腺癌数据brca进行展示(Kataegis在乳腺癌中很常见)。
brca <- system.file("extdata", "brca.maf.gz", package="maftools")
brca <- read.maf(maf=brca, verbose=FALSE)
rainfallPlot(maf=brca, detectChangePoints=TRUE, pointSize=0.6)运行后会输出Kataegis的具体信息,并在图中用黑色箭头进行标记。"Kataegis"定义为包含≥6个连续突变的基因组区段,并且平均突变间距离≤100bp。
## Kataegis detected at:
## Chromosome Start_Position End_Position nMuts Avg_intermutation_dist
## 1: 8 98129391 98133560 6 833.8000
## 2: 8 98398603 98403536 8 704.7143
## 3: 8 98453111 98456466 8 479.2857
## 4: 8 124090506 124096810 21 315.2000
## 5: 12 97437934 97439705 6 354.2000
## 6: 17 29332130 29336153 7 670.5000
## Size Tumor_Sample_Barcode C>G C>T
## 1: 4169 TCGA-A8-A08B 4 2
## 2: 4933 TCGA-A8-A08B 1 7
## 3: 3355 TCGA-A8-A08B NA 8
## 4: 6304 TCGA-A8-A08B 1 20
## 5: 1771 TCGA-A8-A08B 3 3
## 6: 4023 TCGA-A8-A08B 4 3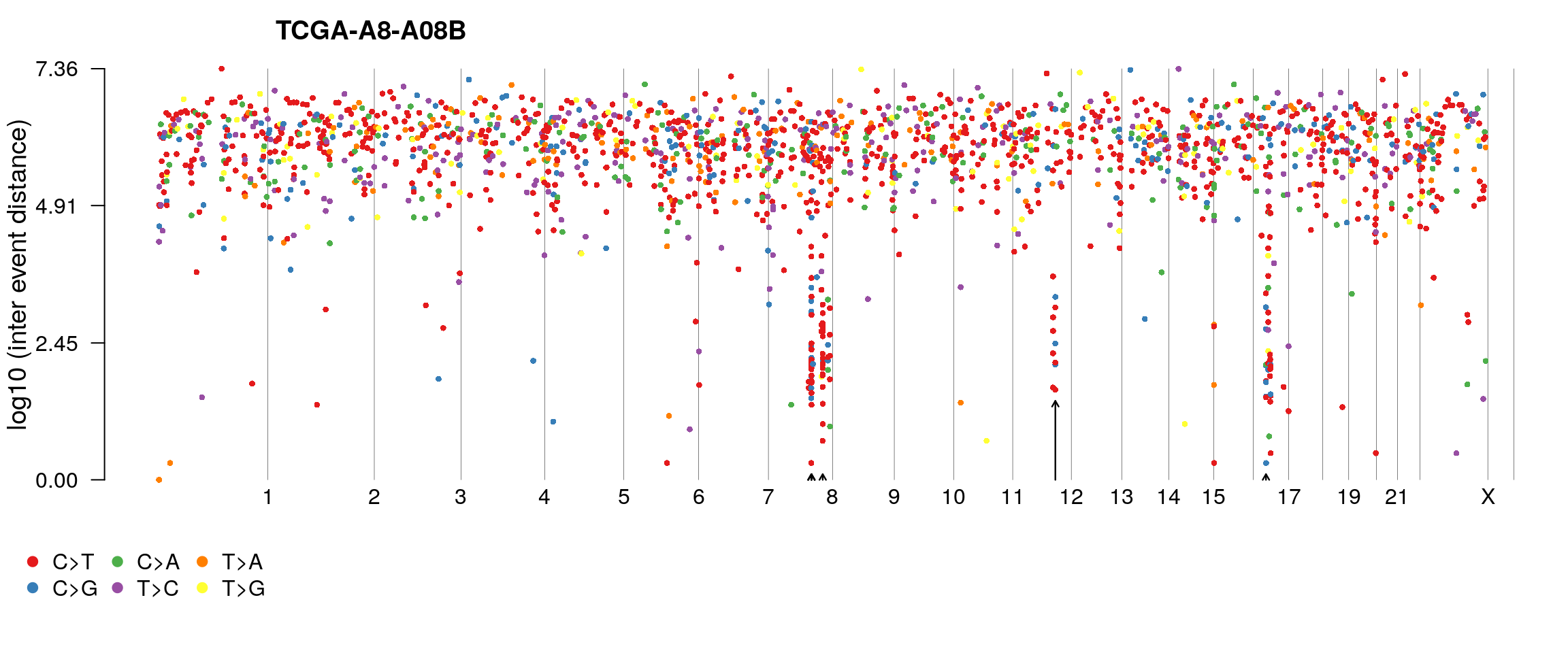
7. 肿瘤突变负荷(TMB)统计以及TCGA cohorts的比较
TMB在很多癌症中与免疫治疗效果以及预后相关,因此也成为这几年的研究热点。Maftools可以使用tcgaComapare函数统计MAF文件的TMB,并和内置的33个TCGA的cohorts进行比较:
luad.mutload <- tcgaCompare(maf=luad, cohortName="Download_LUAD")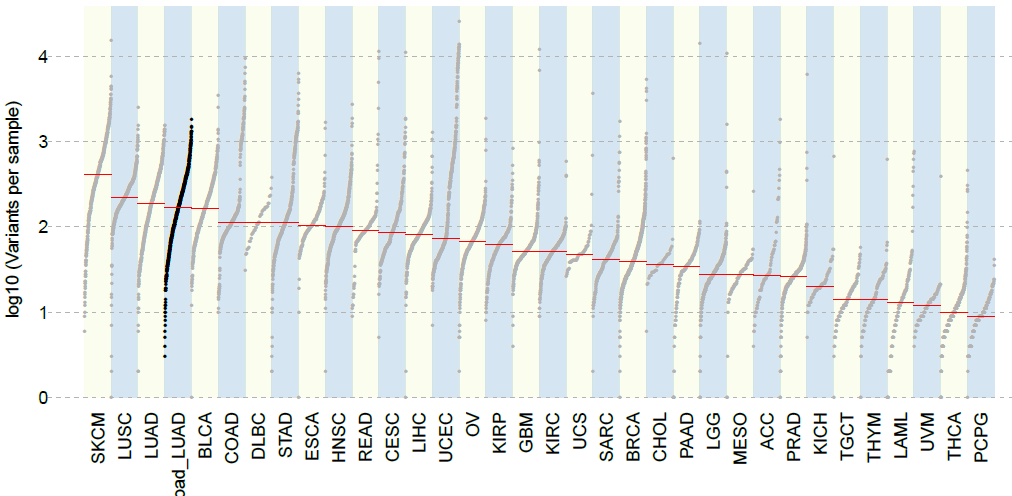
可以看到通过TCGA官网下载的LUAD和这里内置的TCGA-LUAD结果基本上一致(两者Cohort Size有所不同)。
8. Variant Allele Frequency(VAF)可视化
之前已经介绍过VAF的概念了。plotVaf函数可以将VAF最高的几个基因通过箱线图进行展示:
plotVaf(maf=luad)直接运行,会出现提示:
t_vaf field is missing, but found t_ref_count & t_alt_count columns. Estimating vaf..这里程序没有找到默认的t_vaf字段,但会通过t_ref_count和t_alt_count列自动计算VAF。
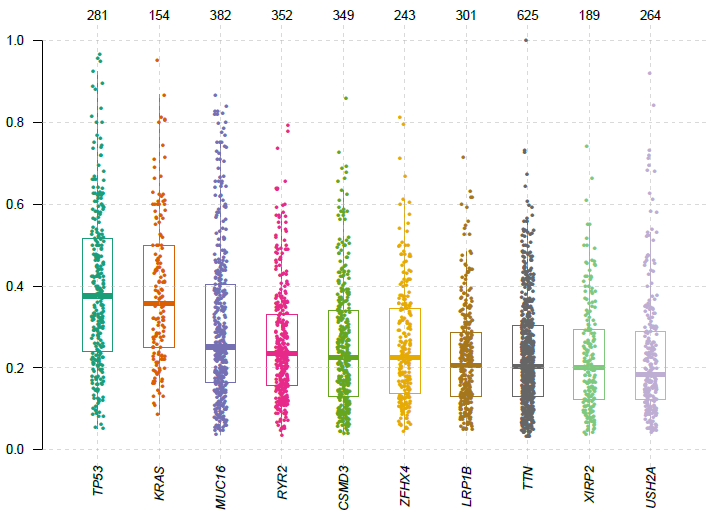
9. 突变基因词云
geneCloud可以通过词云的方式按字体大小展示包含突变基因的样本数。这个函数也可以用来展示GISTC输出的CNV数据。
geneCloud(input=luad, top=10)
 作者:
作者:
我是读入tcga上下载的数据后,然后 直接在R里处理
maf_substr_tsb <- function(laml, end, start = 1){ laml@data$Tumor_Sample_Barcode = substr(laml@data$Tumor_Sample_Barcode,start,end) laml@variants.per.sample$Tumor_Sample_Barcode = substr(laml@variants.per.sample$Tumor_Sample_Barcode, start, end) laml@variant.type.summary$Tumor_Sample_Barcode = substr(laml@variant.type.summary$Tumor_Sample_Barcode, start, end) laml@variant.classification.summary$Tumor_Sample_Barcode = substr(laml@variant.classification.summary$Tumor_Sample_Barcode, start, end) laml@maf.silent$Tumor_Sample_Barcode = substr(laml@maf.silent$Tumor_Sample_Barcode, start, end) laml@clinical.data$Tumor_Sample_Barcode = substr(laml@clinical.data$Tumor_Sample_Barcode,start,end) return(laml) }感谢分享!maftools内基本上也是这个思路处理的数据,比如mafSurvival函数:
if (isTCGA) { clinicalData$Tumor_Sample_Barcode = substr(x = clinicalData$Tumor_Sample_Barcode, start = 1, stop = 12) }请问改好maf格式后是不是还要导出文件,再用read.maf读取maf和clinical data?我直接用read.maf(maf=laml, clinicalData="clinical.tsv")会提示无法强行处理S4数据,如下:
Error in as.character.default(x) :
no method for coercing this S4 class to a vector
如果需要导出的话,请问如何导出maf格式文件呢?我搜了蛮久都没找到解决方法。谢谢!
laml本身就是maf对象,可以直接用
你好,想请教您一个问题。之前拜读了您关于maftools包的使用,非常受益,感谢您的推文。按照您的指导,我从TCGA中下载了LUAD的maf以及临床信息想尝试做一次生存分析。按照您的提示,我直接用excel修改了clinical.tsv中的Tumor_Sample_Barcode以及将Alive和Dead进行了0/1的编码,但使用了mafSurvival指令,仍然出错,提示Time variable is not numeric。尝试了很多办法,仍然未能成功。请问您能否指导一下从TCGA下载了maf以及临床信息后,下一步将怎么处理数据才能使用mafSurvival进行分析呢?冒昧打扰了,非常感谢。
已在《肿瘤变异数据分析和可视化工具maftools:突变的数据分析》中统一回复你的问题
博主,可以分享一下tcga_format.py的脚本吗?谢谢!
正文已更新,我上传到了我的GitHub,可以自取。不过不保证这个脚本可以用在其他癌症的project上
博主您好,假如已经关联了TCGA的临床数据。能否选择特定的临床案例然后用maftools包去分析这些特定案例的突变呢?比如我就想研究淋巴结N3的病人的突变,我应该怎么操作呢?非常感谢
您发的这个Python代码不知道怎么跑出来,麻烦讲解一下
请问下运行python 脚本出现
main()File "tcga-format.txt", line 31, in <module>
File "tcga-format.txt", line 28, in main
handle_maf(sys.argv[1])IndexError: list index out of range
是怎么回事,怎么进行修改呢
果博东方客服开户联系方式【182-8836-2750—】?薇- cxs20250806】
果博东方公司客服电话联系方式【182-8836-2750—】?薇- cxs20250806】
果博东方开户流程【182-8836-2750—】?薇- cxs20250806】
果博东方客服怎么联系【182-8836-2750—】?薇- cxs20250806】