如何去除二代测序数据中的PCR duplication才科学?
最近听闻前公司和另一家公司A因为一个项目中二代测序数据PCR duplication的问题在扯皮。前公司比较两对Paired-end reads的第8~113bp区间内碱基是否完全相同(PE150);而A公司把1~150bp都完全相同的才当作PCR duplication。最终参数的分歧使得两家公司的统计结果相差接近一倍,关乎到数据量是否的满足合同要求。
而前公司这部分QC流程的参数就是我设置的。选择截取reads中间的区域进行比较的原因很简单,因为Illumina测序前面几个碱基和末端的区域通常测序质量较差,测序错误较多。如果要求两对reads完全一致,相当于600bp(150*2*2)不能出现任何测序错误。具体为什么设置成8~113bp我并没有详细去分析和论证,只是因为当时测序都外包给N公司做,而N公司内部QC流程去PCR duplication设置的范围设置的就是这个,我也就顺理成章用了同样的参数。
那么怎样去除二代测序数据中的PCR duplication才是科学的姿势呢?虽然对很多人来说应该是一个无聊的论题,但是我还挺好奇,本文姑且在这里分析一二。
PCR duplication产生的原因和特点
PCR duplication是由二代测序建库过程中的PCR产生的,其特点是插入片段完全相同。因此如果不考虑测序错误,那么测到的序列也应当完全相同。PCR duplication的产生不仅没有提供新的信息,还会影响许多下游的分析。比如通过kmer分析估算基因组的重复率,PCR duplication也会被统计到基因组的重复率中。其他下游分析比如变异检测、利用mate-pair reads进行基因组的scaffolding、Hi-C的数据分析等都会受到影响。
去除二代测序中的PCR duplication的方法
目前有很多现成的软件可以去PCR duplication,我在这里整理了几种常见的软件或方法:
1. 要求全长序列完全相同,以FASTX-Toolkit为代表
FASTX-Toolkit是一个2009年就发布的处理FASTA/FASTQ格式文件的工具包。FASTX-Toolkit中的组件fastx_collapser可用于处理PCR duplication。其原理是将FASTA/FASTQ中的序列进行比较,完全相同的序列只保留一个,和A公司的方式一致。
2. 要求5'端对齐的子序列完全相同,以FastUniq为代表
FastUniq于2012年发表在PLoS ONE上,是一个专门用来快速去除PCR duplication的的软件。相比FASTX-Toolkit仅支持SE reads,这款软件支持PE,并且在运行速度上有较大提高,声称可以在10分钟内处理8700万条reads。
FastUniq支持不同长度的reads序列比较,但是因为是通过归并排序算法(merge sort algorithm)排序后从5'开始逐个位点比较相邻序列的碱基,因此需要两条不同长度的reads在5'端是对齐的。因为需要序列完全相同或者短序列完全被长序列包含,因此和A公司的处理方式也一致。FastUniq的文章中也指出,这种方式由于只基于序列比较,对于杂合或者测序错误引起的SNP是敏感的。
3. 要求截取的部分序列相同,NextClip为代表
NextClip是一款专门针对Nextera MP文库数据的处理工具,于2013年发表在Bioinformatics上。它在处理PCR duplication上使用了kmer-based方法——将一对MP reads的前11个碱基和中间的11碱基共同构成44-mer序列进行比较。
另外有两款比较常用的二代测序数据质控软件会截取部分序列对PCR duplication进行估算——fastp使用了read1的前12bp序列和read2的前32bp序列;FastQC中所有超过75bp的序列会截短成50bp进行比较。两款软件都只是对重复序列进行估算而没有去重复的功能。
另外FastQC的文档中认为用过长的序列会因为测序错误导致低估duplication rate:
Even so, longer reads are more likely to contain sequencing errors which will artificially increase the observed diversity and will tend to underrepresent highly duplicated sequences.
4. Mapping-based method(基于参考基因组比对的方法)
与上面通过reads间比较的de novo法不同,如果有参考基因组,可以通过BWA、Bowtie等比对工具将reads比对到基因组。相同比对位点和方向的reads被认为是可能的duplicates,可以使用Samtools的rmdup或者picard的MarkDuplicates等工具去除或标记重复。
5. 几种方法的总结
这里不讨论基于比对的方法或者unique molecular identifier(UMI)。上面de novo的方法有的用到全长比较,而有的考虑到性能因素只截取少部分序列进行比较(NextClip和fastp最少,一对reads只用到44bp)。序列太短可能会产生假阳性,而序列太长则会对测序错误更敏感。FastQC的文档中就提到,用过长的序列会因为测序错误导致低估duplication rate:
Even so, longer reads are more likely to contain sequencing errors which will artificially increase the observed diversity and will tend to underrepresent highly duplicated sequences.
前面提到两家公司的争论也就在此。一方认为1~150bp过于严格,产生很多假阴性;另一方认为8~113bp过于宽松,会导致假阳性。虽然两条完全随机的序列比较,出现44bp正好相同的概率非常低,只有1/444,但是基因组中并不是完全随机的序列,重复序列会使得问题更复杂。
另外这几个截取部分序列的软件都倾向选择5'端(起始位点为1)。picard MarkDuplicates也是如此,比较的是5'端的坐标。可能是因为越接近3'端,因为InDels导致错位的概率就越高。而reads不同位点测序错误率分布的影响则不是主要关心的问题。
上面这些都只是逻辑上的推理。如何选择合适的序列长度和起始位点,可以使用基于参考基因组比对的结果作为PCR duplication的标准进行评估。
分析方法和结果
1. 测试数据的选取
选取NCBI上公开的一个大肠杆菌E. coli的Illumina HiSeq 4000测序数据和用其组装的基因组,accession number分别为SRR8203406和GCA_002507455.1。因为基于参考基因组比对的方法,基因组重复序列会影响比对的准确率,使用像E. coli这种比较简单的基因组有利于提高分析的可靠性。另外这个基因组组装的连续性也还过得去,41条contigs。
2. 序列比对、标记duplicates
Reads比对和标记duplicates分别使用bwa和picard进行,为减少比对对结果的影响,只保留properly paired reads:
# mapping
fq1=SRR8203406_1.fastq.gz
fq2=SRR8203406_2.fastq.gz
ref=GCA_002507455.1_ASM250745v1_genomic.fna
bwa index ${ref}
bwa mem -t 8 ${ref} ${fq1} ${fq2} > out.sam
samtools view -@ 8 -bS out.sam -o out.bam
# selection of properly paired reads
samtools flagstat -@ 8 out.bam # properly paired: 98.96%
samtools view -@ 8 out.bam -h -f 83 > out.filter.sam
samtools view -@ 8 out.bam -f 163 >> out.filter.sam
samtools view -@ 8 out.bam -f 99 >> out.filter.sam
samtools view -@ 8 out.bam -f 147 >> out.filter.sam
samtools view -bS -@ 8 out.filter.sam -o out.filter.bam
samtools flagstat -@ 8 out.filter.bam # properly paired: 100%
# filtering FASTQ files
python fastq_filter.py \
out.filter.sam \
${fq1} ${fq2} \
fq1.filter.fastq.gz \
fq2.filter.fastq.gz
rm *.sam
# mark duplicates
samtools sort -@ 8 out.filter.bam -o out.sort.bam
java -jar ~/software/picard/build/libs/picard.jar MarkDuplicates \
I=out.sort.bam \
O=out.markdup.bam \
M=markdup.metrics这里只把FLAG为1187、1107、1171、1123的作为duplication(0x400),而2227、2195、2211、2163、2147、2131为嵌合序列的supplementary比对或者比对错误。
3. 真阳性vs假阳性统计
首先统计各指标的值。
| 分类 | 预测阳性 | 预测阴性 |
|---|---|---|
| 真实阳性 | 真阳性TP | 假阴性FN |
| 真实阴性 | 假阳性FP | 真阴性TN |
这里我们把通过picard MarkDuplicates得到的重复序列作为set1。把通过de novo序列比较预测的重复序列作为set2;以重复一次为例子,如果read对A和read对B经过比较相同,两者都统计进去;同理,如果read对A被标记为重复,根据picard MarkDuplicates的原理,相同5'坐标和方向的read对B也统计进去。得到set1和set2之后,以set1为标准衡量set2的结果,全集为setU:
TP = set1 & set2
FN = set1 - set2
FP = set2 - set1
TN = setU - (set1 | set2)
然后求真阳性率TPR、假阳性率FPR、精确率Precision和F1 score:
真阳性率TPR = Sensitivity = Recall = TP/(TP+FN)
假阳性率FPR = 1-Specificity = FP/(FP+TN)
精确率Precision = TP/(TP+FP)
F1 = 2*Precision*Recall/(Precision+Recall)
通过坐标从1开始,步长2;reads截取长度从6bp开始(双端共6*2=12bp),步长2进行穷举,计算上述4个指标,最终得到统计结果stat.txt:
python result_stat.py out.markdup.bam fq1.filter.fastq.gz fq2.filter.fastq.gz4. 结果分析
(1) TPR、FPR、Precision、F1随reads截取长度的变化
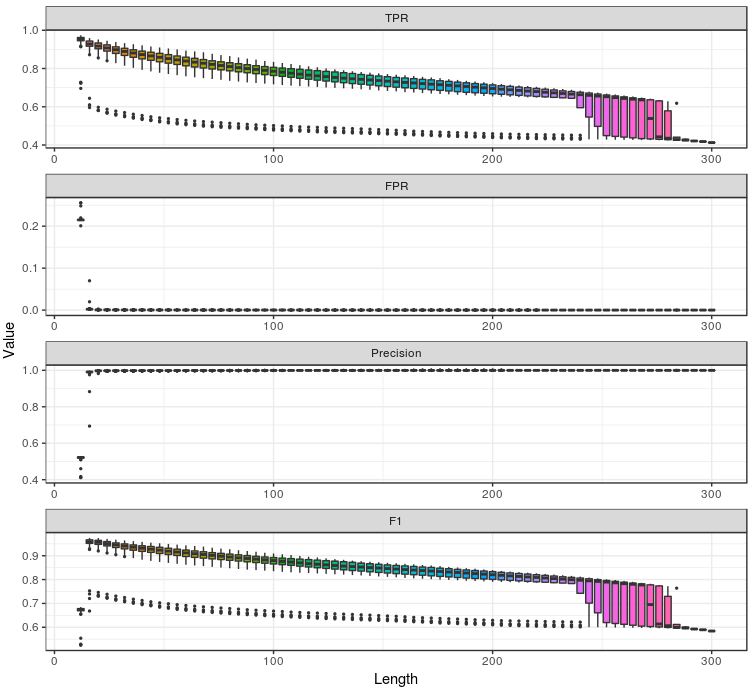
- TPR:从整体上看TPR随着长度增加而逐渐减小,可能的原因是序列越长,测序错误越多,导致真阳性率下降。当使用全长序列进行分析时,TPR仅有0.41。
- FPR:除了截取长度为12bp(read1和read2各6bp)时FPR高达0.20~0.26,基本上FPR都处于很低的水平,说明只要read1和read2分别取大于等于8bp序列在本例中就能保证非常好的特异性。
- Precision:同理,除了截取长度为12bp的情况,Precision都相当高。
- F1 score:F1是平衡Precision和Recall(TPR)的综合评价指标,由于除了截取长度在12bp以外Precision都极高,因此F1的变化趋势和TPR基本保持一致。
- 较短截取长度(12、16、20bp)会导致FPR和Precision在接近3'端出现很大变化,在图中体现为离群点。
(2) TPR、FPR、Precision、F1随位点的变化
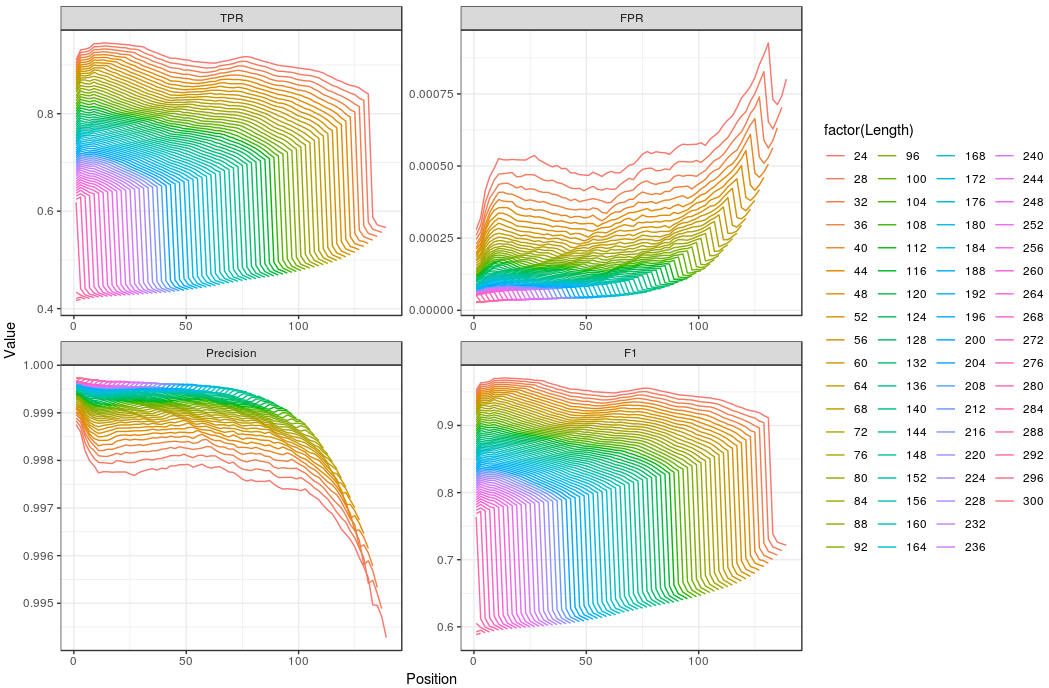
这里因为较短截取长度在接近3'端的FPR/Precision变化太大,所以去除了12、16、20bp的情况。
- 不同截取长度之间的TPR、FPR、Precision、F1随位点变化趋势非常一致。
- 由于Precision极高,F1和TPR的变化趋势基本一致。
- TPR、F1在5'端附近先增加后下降,而在3'端急速下降。这可能和5'端以及3'端较高的测序错误率有关。
- TPR、F1、Precision都大体呈现随位点下降趋势,FPR则随位点升高。可能是因为越接近3'端因为InDels导致错位的概率越大。
- FPR在5'端和3'端上升,Precision相反,但总体上变化并不大(Y轴)。
(3) 按照F1排序的Top 5
| TPR | FPR | Precision | F1 | Range | Length |
|---|---|---|---|---|---|
| 0.959334100718 | 0.00212222364236 | 0.991087803814 | 0.97495247033 | 13-20 | 16 |
| 0.958972438393 | 0.00213351446829 | 0.991037464688 | 0.974741320595 | 15-22 | 16 |
| 0.958140798631 | 0.00212944977095 | 0.991046692023 | 0.974315989128 | 17-24 | 16 |
| 0.95184383531 | 0.000592090911924 | 0.997477813027 | 0.97412667427 | 15-24 | 20 |
| 0.951706146607 | 0.000597058875334 | 0.997456337318 | 0.974044324192 | 13-22 | 20 |
(4) 计算两家公司的方法以及几种现有软件的指标
| Method | TPR | FPR | Precision | F1 | Range | Length |
|---|---|---|---|---|---|---|
| 前公司 / N公司 | 0.7103396872 | 7.722924938e-05 | 0.9995582514 | 0.8304892118 | 8-113 | 212 |
| A公司 / FastUniq | 0.4125667561 | 2.709798224e-05 | 0.9997330818 | 0.5840921643 | 1-150 | 300 |
| NextClip | 0.8638864564 | 0.0001151664245 | 0.9994583917 | 0.9267405003 | 1-11, 76-86 | 44 |
| fastp | 0.8698327725 | 0.0001878793435 | 0.9991227700 | 0.9300057806 | 1-12; 1-32 | 44 |
以F1 score作为衡量标准,方法优劣排序如下:
fastp ≈ NextClip > 前公司/N公司 > A公司
这里没有比较FASTX-Toolkit和FastQC,因为这两款软件都只支持单端。
结论 & 讨论
- 过长的截取片段的确可能因为测序错误导致重复比例被低估。按照A公司策略选取1-150全长进行分析,TPR只有0.413,按照前公司和N公司的策略选取8-113时TPR为0.71。这可以解释为什么两家公司结算结果有如此大的差异。
- 在本例中,仅选取片段>=16bp就能得到不错的特异性。F1值最高时截取的序列范围为13-20,共(20-13+1)*2=16bp。
- 5'端和3'端测序错误可能导致重复比例被低估(TPR下降),5'端的影响并不是特别大,但3'端附近的TPR急速下降。
- 本例使用
picard MarkDuplicates作为标准,为了防止错误比对选择了基因组重复较低的E. coli,并且只用properly paired reads构成的subset进行分析。 - 对于复杂基因组测序结果,重复序列比例应该要高很多,因此FPR应该比本例的要高,为了保证方法的稳健性,还是应当提高截取序列的长度,仅使用16bp可能不够。而TPR应该影响不大。
代码获取
本文分析中数据下载、比对、过滤、运行picard MarkDuplicates、以及TPR、FPR、Precision、F1计算的代码可以去我的GitHub查阅:
参考资料
- FASTX-Toolkit: https://github.com/agordon/fastx_toolkit
- FastUniq: https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0052249
- NextClip: https://academic.oup.com/bioinformatics/article/30/4/566/202704
- fastp: https://academic.oup.com/bioinformatics/article/34/17/i884/5093234
- FastQC: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/Help/3%20Analysis%20Modules/8%20Duplicate%20Sequences.html
- Picard: https://broadinstitute.github.io/picard/command-line-overview.html#MarkDuplicates
 作者:
作者:
您好,文章中FN = set2 - set1的定义错了
感谢指正!文章里FN和FP写反了,已改正,代码写的是对的。