Oxford Nanopore碱基识别(basecalling)软件性能大比拼
本论文除了评测了ONT开发的4个basecaller以及1个第三方的basecaller以外,还对不同方法的自训练模型进行了较为系统的评估,是目前最详细的对Nanopore碱基识别软件(basecaller)的横向评测的研究,非常值得仔细阅读。文章于今年2月在预印本在线期刊bioRxiv上发表,作者是澳大利亚Monash University的Ryan Wick。
研究背景
1. ONT识别碱基的原理
- 膜两端存在电压差。
- 通过nanopore的单链DNA分子的核苷酸会产生不同的电阻。
- 测量的电流信号随时间的变化可以识别对应的碱基。
2. 其中的难点
- ONT为单分子测序,因此噪音信号和随机序列对basecalling的准确性影响大。
- 一次通过孔有多个碱基(以R9.4为例大约5个碱基),因此存在大量可能的状态(45=1024)。
- 由于碱基修饰的存在,会导致情况更复杂(假设把5mC当作第5类碱基,就会有55=3125种可能的状态)。
3. 目前Nanopore basecaller的现状
- ONT公司和独立的研究者都在开发basecalling方法。
- 如今的basecaller都使用神经网络解决这个问题,需要使用真实数据训练。因此basecaller的效果会受到训练集的影响——是否有碱基修饰、序列motif存在。
4. basecalling的准确性
- basecalling准确性评估通常关注两个方面:原始read水平的准确性和consensus(一致性序列)的准确性。
- 其中两者可能相关,也并不一定如此,这取决于错误是随机错误还是系统性错误。
- 通常我们更关注consensus的准确性,当然在使用低深度测序的应用中,read的准确性很重要。
5. 研究内容和目的
基于ONT的R9.4对多种basecalling软件进行性能上的比较,并探索了模型训练对basecalling准确性的影响。本研究可能对希望最大限度挖掘ONT测序信号的研究者提供指导,以及帮助读者决定是否有必要用新的软件或自训练模型重新对信号数据进行re-basecalling。
研究方法
1. 受测的basecaller
包含4个ONT开发的basecaller(测试了所有兼容R9.4的版本),以及一个第三方开发的:
- Albacore:通用的basecaller,通过CPU运算,目前由于Guppy性能更优已停止开发。
- Guppy:Albacore的继任者,通过GPU提高了运行速度(包含最新的flip-flop)。
- Scrappie:包含Scrappie events和Scrappie raw。“技术先驱”,总是尝试使用新的方法,随后加入到Albacore和Guppy中。
- Flappie:最近替代了Scrappie,使用CTC(Connectionist temporal classification)识别碱基
- Chiron:第三方basecaller,相比ONT的basecaller使用了更深层的神经网络。
- 其他不再开发的软件排除在测试之外:Nanonet,DeepNano,basecRAWller。
2. 模型训练
为研究训练集对basecaller(这里主要是对Guppy)性能的影响,使用50个基因组构建了针对Klebsiella pneumoniae(肺炎克雷伯氏菌)的训练集——包括30个K. pneumoniae、10个Enterobacteriaceae(肠杆菌科)的其他物种,以及10个Proteobacteria(变形菌门)物种。使用ONT的神经网络训练工具集Sloika进行训练。Illumina reads用SKESA组装contig。
流程如下:
- 原始ONT reads,在fast5水平(基于电信号)进行trimming、丢弃短reads。
- 使用Albacore进行basecalling,再根据长度、比对illumina contig的完整性,sliding window的质量进行过滤。
- 多次针对“原始信号对参考基因组比对”的质量进行过滤(参照Sloika的训练流程)。
- 每个基因组选20条reads作为验证集,剩下的作为神经网络的训练集,使用Sloika训练。
3. 用于评估basecaller的read数据集
- DNA来源于K. pneumoniae,选该物种做评估的依据是高ONT reads产量、无质粒使得更易于组装。
- 物种与训练数据为相同物种,但是lineage不同(isolate INF032)。
- 数据为原始DNA,因此包含碱基修饰。最主要类型为Dcm甲基化。
- 除了ONT reads,还测了illumina reads用于混合组装构建INF032的参考基因组(Unicycler)。
- ONT reads通过拆barcode、Guppy basecalling、minimap2比对、长度筛选后选取的子集用于作为评估basecaller的数据集(后称评估集)。
- 准备了另外9个基因组的数据集作为附加数据集用于细致评估不同版本的Guppy。
4. Read准确性以及consensus准确性评估
为了评估各basecaller的运行速度,basecalling在同一台电脑上运行。
Read准确性评估方法:
- Read准确性评估通过minimap2比对到构建的INF032参考基因组,并计算“BLAST identity”,未比对上的记作0%。
- 将identity的中位数定义为每个basecaller的总体read准确性。
Consensus准确性评估方法:
- 使用Rebaler生成consensus基因组序列。
- 将consensus基因组序列打断成10kb的片段,与上述ONT reads相同方法评估准确性。
- 使用NUCmer比对reference鉴定consensus序列的错误类型。如果在Dcm甲基化motif(CCAGG或CCTGG)中出现,就分类为“Dcm”;如果序列在≥3个连续碱基的位置出现1个碱基的增加或缺失则定义为“homopolymer insertion”或“homopolymer deletion”;其他的则对应分类为“insertion”、“deletion”、“substitution”。
5. 关于Polishing:
本研究主要关注于评估basecaller的性能,因此polishing在主题之外。但很多用户做纯Nanopore组装后会用原始read信号运行Nanopolish纠错,所以引入了另一个问题:评估如果用Nanopolish的话,basecaller的选择还有那么重要吗?为了回答这个问题,文章对Rebaler得到的consensus序列又做了Nanopolish并评估了consensus的准确性
结果和讨论
1. 默认模型下的性能比较
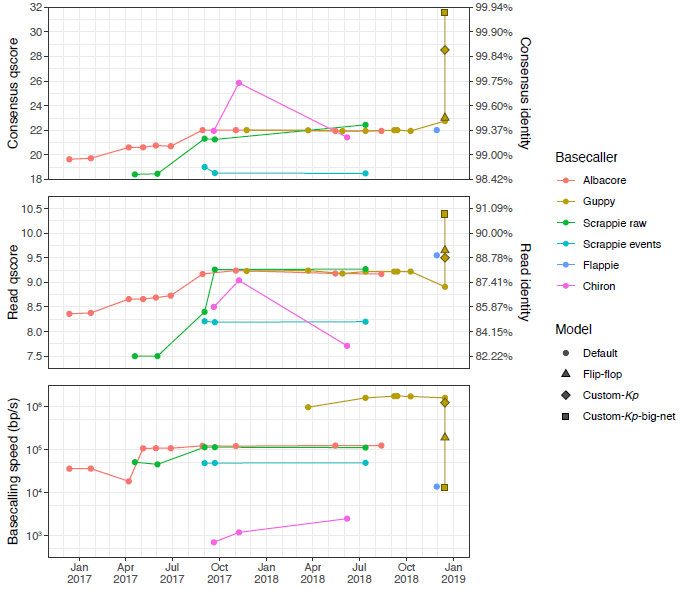
准确性的比较:
- Albacore两次read准确性和consensus准确性都比较明显的提升是在2017年4月(v1.0.1)和2017年8月(v2.0.1)。然而后续的版本中准确性没有变化,受测的最新版read准确性为Q9.2,consensus准确性为Q21.9(v2.3.4)。
- Guppy准确性一直比较恒定,于Albacore的大部分版本近似。最新版的Guppy(v2.2.3,2019年1月)read准确性较差(Q8.9),但是consensus准确性较好(Q22.8)。但是这个版本中引入的“flip-flop”有相似的consensus准确性但是好得多的read准确性(Q9.7)。
- Scrappie events是受测basecaller中最差的一款,但是Scrappie raw表现更好,在最新一版中(v1.4.1)已经和Albacore表现相似(read和consensus准确性分别为Q9.3和Q22.4)。Scrappie的继任者Flappie(2018年11月)read准确性更好(Q9.6),但是consensus准确性较差(Q22.0)。
- 相比ONT开发的basecaller,第三方的Chiron的read准确性很差。但是Chiron v0.3是所有basecaller中默认模型下consensus准确性最高的一款(Q25.9)。最新的Chiron(v0.4.2)表现下滑。
运行速度的比较:
- Albacore和Guppy准确性相似,但是Guppy因为使用GPU运算加速所以快得多(1,500,000 bp/s vs 120,000 bp/s)。
- 虽然Chiron也使用GPU加速,但是却是所有basecaller中最慢的一个(~2,500 bp/s)。完成MiniION典型产量(10 Gb)的basecalling需要大约一个月时间,除了很小的数据集,否则基本上没法用。
- Flappie运行速度也很慢(~14,000 bp/s)
2. 自训练模型下的性能比较
- 相比默认模型,通过50个基因组训练的custom-Kp模型在read准确性上有中等提升(Q9.5),在consensus准确性上有很大提升(28.5),说明使用分类特异性的训练数据对提高准确性有帮助。此外运行速度方面和默认模型相比相似。
- custom-Kp-big-net模型在read准确性和consensus准确性上表现更好(分别为Q10.4和Q31.6)。更复杂的神经网络可以提高准确性,但是运行速度下降了。custom-Kp-big-net模型不能使用GPU是因为使用到了没有在Guppy中预编译的神经网络层,因此只能使用CPU运行,导致比Guppy的默认模型和custom-Kp模型慢得多(~13,000 bp/s)。
此外还使用远源的9个基因组的附加数据集详细评估Guppy各模型的表现:
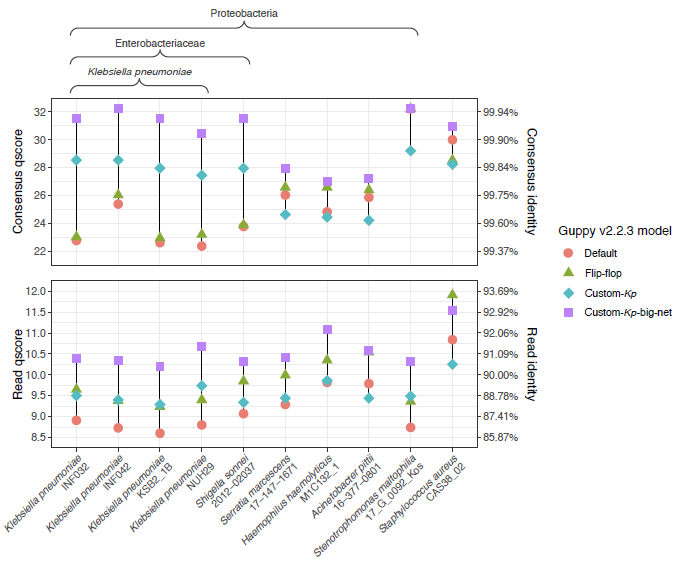
- flip-flop模型在read和consensus准确性的表现比默认都要好(质量值分别+0.71和+0.36)。
- custom-Kp模型比默认模型在肠杆菌科基因组的表现要好得多(质量值分别+0.63和+4.72),但是在非肠杆菌科基因组中没有帮助(0.00和-1.64)。分类特异性的准确性提高可能是因为custom-Kp模型提高了肠杆菌科中Dcm甲基化motif的basecalling准确性。
- custom-Kp-big-net模型相比custom-Kp模型不是分类依赖的,而是在所有的基因组中都有提高(read和consensus质量值分别平均提高了1.01和3.15)。在绝大多数情况,custom-Kp-big-net模型都得到了最准确的reads和consensus序列,除了S. aureus中flip-flop的准确性更好。
- custom-Kp和custom-Kp-big-net模型都没有用到Guppy中新的神经网络架构flip-flop模型。理论上同时基于flip-flop架构和自训练的模型可以同时获得两者的优势,但是目前版本的Sloika(v2.1.0)还不能使用flip-flop架构进行模型训练。
3. Consensus错误概述
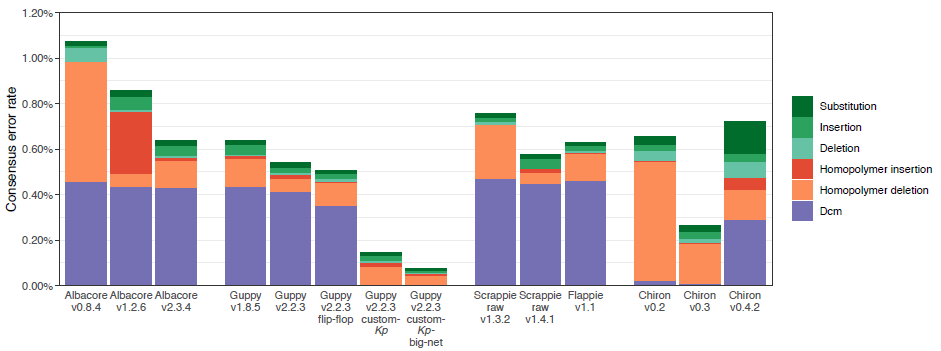
Dcm甲基化位点错误:
- 所有ONT的basecaller在使用默认模型时在Dcm甲基化位点的准确性都很差,使得这类错误在consensus序列总错误中占了很大比例(占参考基因组的~0.4%)。说明使用缺乏Dcm甲基化的数据训练的模型没有学会可靠地call出这些位点的碱基。
- 相反,使用自训练模型的几乎没有Dcm错误(~0.002%),因为训练集中包含Dcm甲基化位点。
- 由于Chiron v0.2和v0.3基于包含Dcm修饰的E. coli reads训练,因此Dcm错误很少(<0.025%)。但是Chiron v0.4.2显示出0.29%的Dcm错误(整体错误也更多),说明它与之前版本使用了不同的数据集进行训练。
Homopolymer长度错误(包含homopolymer insertion和deletion):
- Dcm motif之外,homopolymer的长度错误是最主要的错误类型。
- Albacore随着新版本的发布在homopolymer长度错误有着最显著的改善(从v0.8.4的0.53%下降到v2.3.4的0.13%),显示出ONT在这方面有明显的进步。
- 最新的Guppy(v2.2.3)有更进一步的改善,使得Homopolymer长度错误下降到0.07%。
- 自训练的custom-Kp模型相比默认模型表现较差(0.10%),而custom-Kp-big-net表现较好(0.05%)。
4. Nanopolish的表现
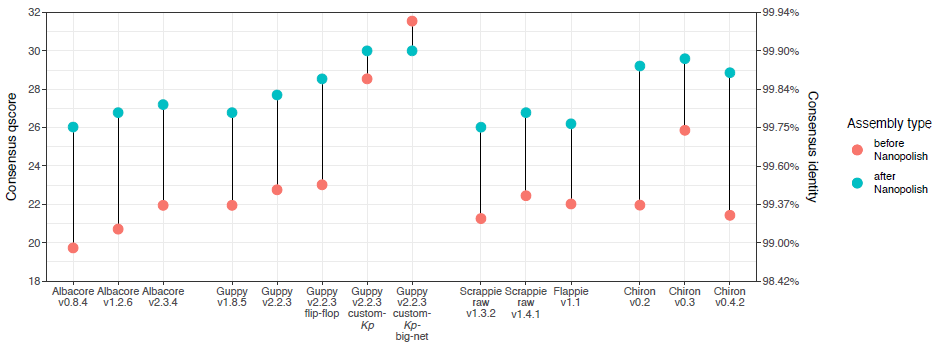
- 除了custom-Kp-big-net以外,Nanopolish在所有版本和Guppy的各模型中都提高了consensus的准确性。
- Nanopolish前的consensus准确性和Nanopolish后的准确性相关(R2=0.580),说明即使用了Nanopolish,basecaller的准确性依然重要。
- Nanopolish可以统计Dcm甲基化(使用
--methylation-aware参数),但通常仅能纠正~70~80%的Dcm错误。据此,Nanopolish前的Dcm错误就成了Nanopolish后准确性的最好预测值(R2=0.809)。 - Guppy v2.2.3的组装结果多轮Nanopolish只有很小的准确性提高(4轮后从Q27.5提高到Q28.3)。
- 各版本组装的基因组中单碱基替换最少仍有337个(使用Guppy v2.2.3、custom-Kp-big-net模型外加Nanopolish),这些假阳性在食物传播和其他传染疾病等研究应用中是不可接受的。虽然使用定制的策略进行ONT reads的SNP calling可以降低假阳性,但是基于组装的序列比对的basecalling准确性亟待很大提高。
结论
使用Guppy v2.2.3并通过大部分来自同一物种的序列进行自训练的方式在read和consensus序列都有最高的准确性。这似乎主要是因为可以准确处理Dcm甲基化。由于不同分类有不同的DNA修饰模式,因此更普适化的结论是:当使用拥有相似DNA修饰的同一个物种或者足够近缘物种的原始DNA(未经过扩增)进行训练后的basecalling,原始DNA的准确性更高。
对于绝大多数basecaller,用于模型训练的DNA的分类和DNA类型(扩增与否)的完整信息都没有公开,我们鼓励开发者在这方面更加透明并如果可能的话,考虑提供更多样化的训练模型(扩增的、人类的,为扩增的E.coli DNA等)。这样用户可以选择一个与他们物种更接近的模型。而对于拥有足量训练数据、带GPU的高性能计算机、以及计算机技能的用户,我们建议通过自训练模型最大可能提高basecalling的准确性。文章中的custom-Kp-big-net说明当使用更多层的神经网络,还能进一步提高准确性。
近几年ONT测序技术已经在产量和准确性上有了长足的进步,但是文章的结果显示仍旧有很大的进步空间。测试的所有basecaller、模型和基因组中,最好的consensus准确性是Q32.2(99.94% identity)。这相当于在一个5 Mb的基因组中平均存在~3000个错误。其中很多错误是单碱基替换,这会使得可能产生很多假阳性SNP而成为暴发调查的主要障碍。为了得到完美的细菌基因组组装,consensus准确性需要几个数量级的提高,比如Q70(1个错误/10 Mb)。可能需要在这些方面提高:技术和化学试剂上的变化,basecalling的进步,组装后polishing工具的开发。在实现这些目标之前,混合组装或使用Illumina reads进行polishing对需要高准确性序列的研究者来说仍旧有必要。
附录
相关链接:
- bioRxiv链接:https://www.biorxiv.org/content/10.1101/543439v1
- 文章中用到的代码脚本:https://github.com/rrwick/Basecalling-comparison
- 作者GitHub主页:https://github.com/rrwick
推荐Ryan Wick的其他比较有名的项目:
 作者:
作者:
暂无评论