跟风做课题有风险,你研究的eRNA靠不靠谱?
增强子RNA(Enhancer RNA,eRNA)近期好像因为Nature Communications的一篇文章又火了一下,作者还甩出了一个叫eRic的数据库[1],里面不仅整理好了TCGA各类癌症中鉴定到的eRNA,表达量,还关联上了病人的生存,甚至还有药物处理后的变化。临床和实验基础的课题组看到这篇文章说不准欢呼雀跃快马加鞭做起了功能机制研究。不过在做新方向课题之前,有没有好好想过,别人的分析真的靠谱吗?
简单介绍eRNA
先做一点简单eRNA背景介绍:(1)激活的增强子被发现通常会转录产生RNA,称作eRNA;(2)eRNA有个特征就是会双向转录(bidirectional transcription);(3)根据双向转录这个特征,FANTOM5通过对808个人类组织或细胞(目前已经更新到了1829种)进行CAGE-seq(Cap Analysis of Gene Expression)鉴定了目前最“靠谱”的eRNA/激活的增强子数据集,并于2014年发表了一篇Nature [2]。为什么这里“靠谱”要用引号,本文的最后一节会有解释。
eRNA的pan-cancer研究
然后能下载到TCGA原始RNA-seq数据的大佬们(如果你也是能下载到TCGA原始数据的大佬,可以合作,请联系我!)就开始想把这个研究方向带到癌症的研究中,因此以FANTOM5的enhancer数据集为基础,进行了pan-cancer eRNA分析。先后在2018年和2019年分别发表了一篇Cell[3]和这篇NC[4]。都是大文章呀,是不是令人心动!
Cell这篇文章比较乖,就是用的FANTOM5的的数据集,然后还做了一系列筛选,看着挺严格的,而且表观遗传的那些features也挺符合预期,但是不是双向转录没法用TCGA的RNA-seq数据验证,因为常规的RNA-seq建库测序方法不能准确识别转录起始位点(Transcription Start Site,TSS),非特链特异性建库也没法判断转录方向。
而最近的这篇NC就“不那么乖”了,除了FANTOM5的数据,还用上了ENCODE和RoadMap的enhancer数据集,毕竟差不多的分析想要发好文章,总要有点新意不是?不过这俩数据库是通过H3K4me1和H3K27ac这两种组蛋白修饰鉴定的enhancer,按照FANTOM5 enhancer那篇文章的意思,由CAGE鉴定的转录的enhancer比由组蛋白修饰或DNase I超敏位点(DNase I hypersensitive site,DHS)鉴定的非转录的验证率高得多:
In summary, active CAGE-defined enhancers were much more likely to be validated in functional assays than untranscribed candidate enhancers defined by histone modifications or DHSs.
到这里已经有点忍不住开始吐槽了,虽然NC这篇文章要求至少有两个数据库支持,听起来似乎很靠谱,可是没觉得ENCODE和RoadMap这两个用相似方法的不是一伙的吗?另外用TCGA这类常规RNA-seq数据做这个分析有个很大的局限,文章中也做了说明:
Our method may only detect a subset of polyadenylated eRNAs at their steady state since TCGA and CCLE only included the poly(A) RNA-seq.
这还是值得敬佩的,毕竟按FANTOM5那篇nature中说,eRNA大约90%都是无poly(A)的,那这个subset,可能就只有~10%了。当然,这不代表10%的eRNA只能鉴定10%的enhancer。另外这点对做实验的人来说也无所谓,只做poly(A)的subset也是没问题的,只要找到的靠谱就行了。那eRic数据库中的结果到底靠不靠谱呢?我这里根据自己的研究方向选择肺腺癌为例(LUAD)。
eRic数据库中的eRNA真的靠谱吗?
首先从eRic数据库[1]下载文件eRNA.m1.csv,从中筛选出LUAD的eRNA,并转换为BED格式进行可视化。
eRNA之间的overlap甚至完全重合
首先初读NC这篇文章,就会让人有个困惑的地方。里面过滤了不少和别的region的overlap的eRNA,比如和已知coding region还有lncRNA,那eRNA和eRNA之间呢?如果两个eRNA之间挨得比较近,他们的flanking region会不会出现overlap呢?答案是会有,我们用IGV来看看会比较直观,比如文章拿了出来详细讲了的NET1相关的eRNA NET1e:
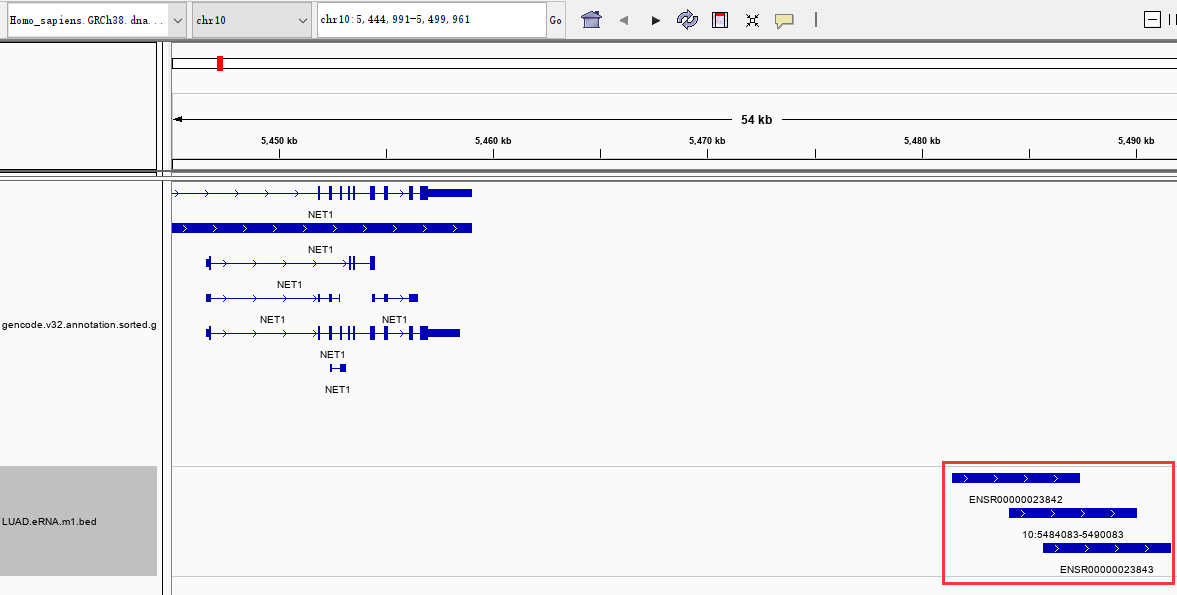
Enhancer的region有重叠是可以理解的,在不同的组织和细胞中可能会有靠得很近的enhancer被激活。但是这种类型的enhancer,不用CAGE-seq,而只通过普通的RNA-seq应该是不能准确区分的,因为存在TSS在某个enhancer,但是转录区会跨越另一个enhancer的可能。
另外更让人摸不着头脑的是:坐标和表达量都完全相同但是用了不同ID的eRNA:
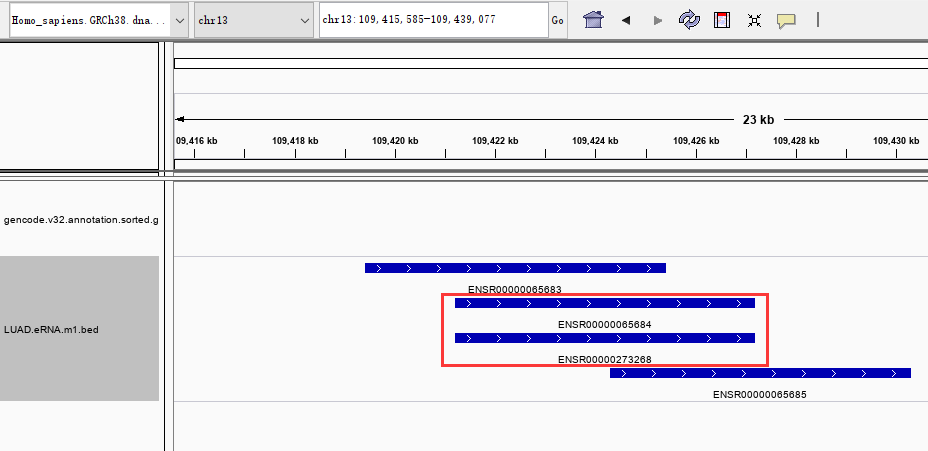
LUAD坐标和表达量完全相同的eRNA共有两处:
,cancer_type,id,location,chromosome,chr_start,chr_end,mean_exp
6513,LUAD,ENSR00000065684,13:109421200-109427200,13,109421200,109421200,20.736656070514
7173,LUAD,ENSR00000273268,13:109421200-109427200,13,109421200,109421200,20.736656070514
6729,LUAD,ENSR00000142306,21:38353300-38359300,21,38353300,38353300,1.71989303454777
7239,LUAD,ENSR00000300368,21:38353300-38359300,21,38353300,38353300,1.71989303454777所以你敢想象和另一个课题组研究了两个名字不同但是部分相同甚至完全相同的eRNA吗?
eRNA还可能和coding gene有overlap
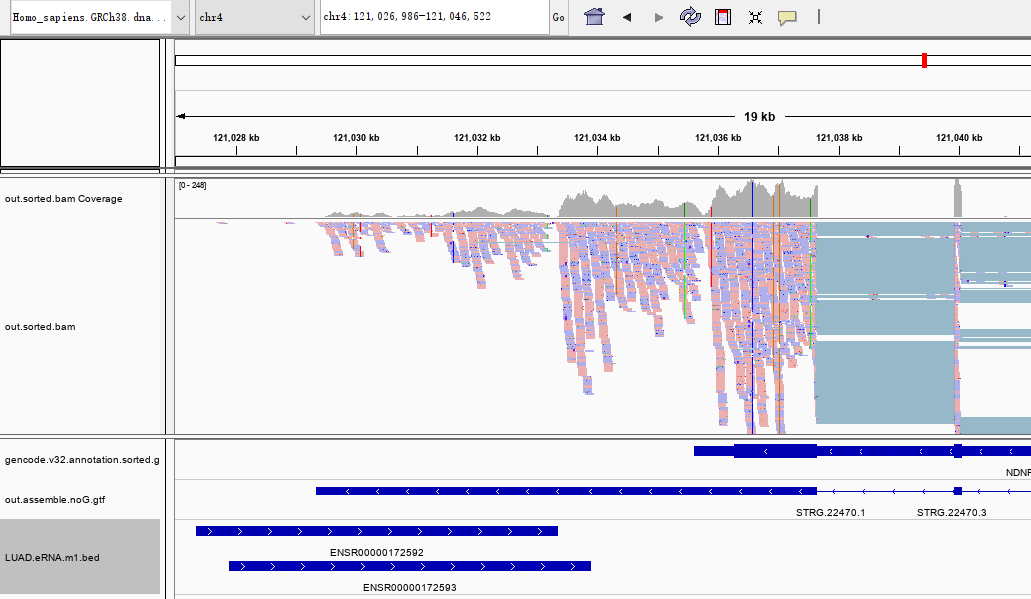
我还把肺腺癌里eRNA按照表达量做了排序,比如表达量最高的这个,距离基因非常近。下载了一个肺腺癌病人的RNA-seq数据,用HISAT2比对,并用StringTie进行组装,发现这个eRNA虽然没有和注释的coding gene有overlap,但是和StringTie的组装结果的3'有overlap。检测到eRNA的表达,可能只是NDNF基因3'末端的可变多聚腺苷酸化(alternative polyadenylation,APA):
eRNA说不准还有可能和一个已知的反转录转座子有overlap
坐标chr2:148253179-148259179的eRNA ENSR00000124270与坐标chr2:148253985-148259246的LTR HERVL18-int存在overlap,其表达也可以在LUAD A549细胞系中得到验证:
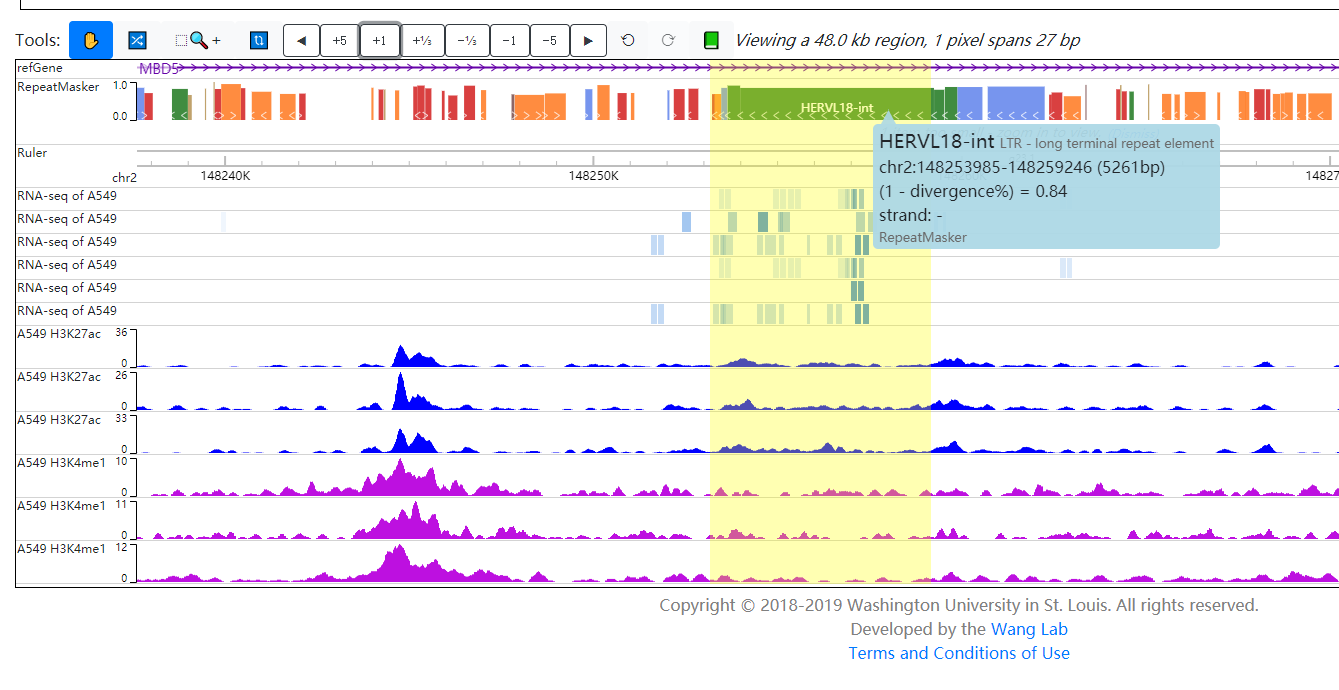
和已知转座子有overlap的情况找到很多,我也就不一一贴出来了。这个结果和Nature文章中的描述有一些矛盾:
repeat regions are generally under-represented in CAGE-defined enhancer regions compared to randomly selected genomic regions
当然,转座子中存在alternative enhancers我认为也是可能的,但是与已知反转录转座子overlap会增大假阳性的风险,毕竟这只是RNA-seq数据做的分析。
文章分析的eRNA靶基因可不可靠?
文章里找靶基因的准则是距离在≤1Mb以内,Spearman相关系数≥0.3,FDR<0.05。要问我觉得靠不靠谱,直接甩一张文章里的图:
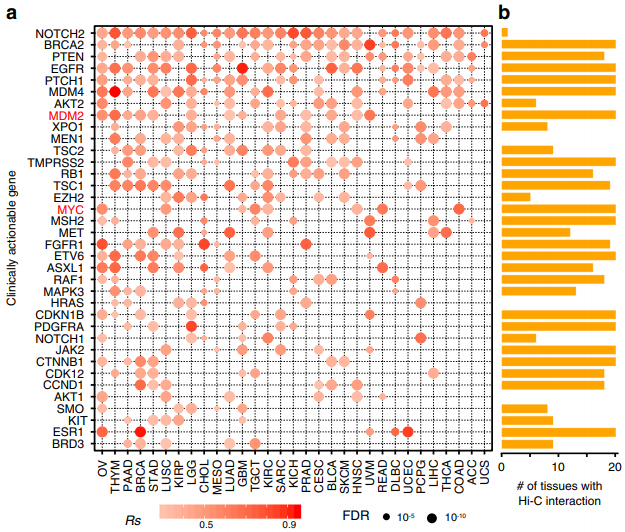
Hi-C应该算是很直接的证据了,因为包含非特异性互作,甚至相比Polymerase II的ChIA-PET还有更多假阳性,但NOTCH2这个基因的验证率是怎么回事?
写在最后
此外还有其他问题,比如在上一篇文章《BED文件也有可能是1-based坐标系?》中也提到过的,FANTOM5的BED文件可能是1-based坐标系,这些pan-cancer的研究是否注意到了这点,如果没注意到对结果是否又会有影响呢?分析到这里,如果是我,我是不敢随便用里面的数据去做功能研究了,会慎之又慎,否则到最后可能做出功能了也不知道自己做的是个什么鬼东西。即便要做,也会反复用IGV多检查检查,再加上FANTOM5里对应细胞系的CAGE-seq、以及其他数据库中ChIA-PET或者Hi-C的数据进行验证。
平时我可能是懒得特地去做这种分析还写一篇文章的,但是本来也正是因为NC这篇论文想了一个拳打Nature,脚踩Cell和NC的eRNA相关的课题(误)——FANTOM5中LUAD只用了3种细胞系的CAGE-seq,而我手头又有26种LUAD细胞系的TSS-seq的数据(一种类似CAGE-seq的测序技术),可能可以增加不少新的LUAD特有的激活enhancer。
一上来就大张旗鼓分析数据成本和风险都比较高,所以我直接拿FANTOM5的数据先做些简单的分析——CAGE peaks随样品数量的饱和曲线,以及现有的3个LUAD细胞系对结果的贡献几何。最终理想很丰满,现实很骨感,我直接拿FANTOM5的数据做了些简单的分析,发现用了1829个细胞或组织样品的效果不得不说真是太好了,能鉴定到的CAGE peaks已经基本饱和:
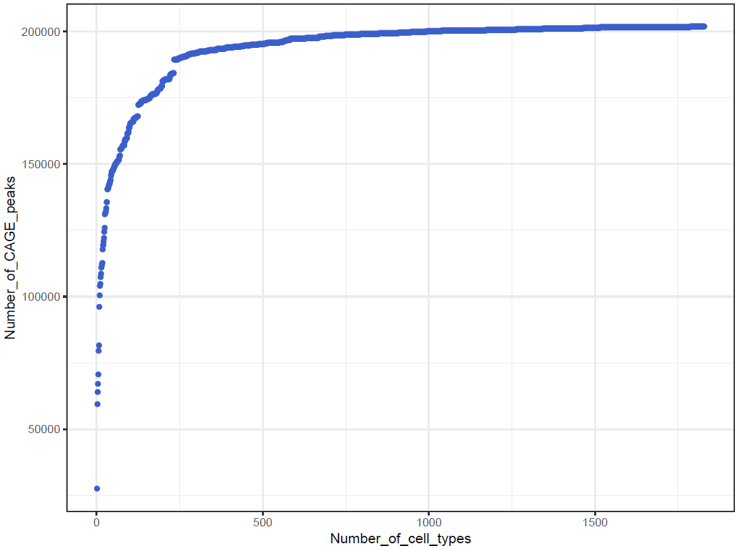
并且即使一个LUAD的样品都不用,也只会少3个特有的CAGE peaks,特有的激活enhancer只会更少:
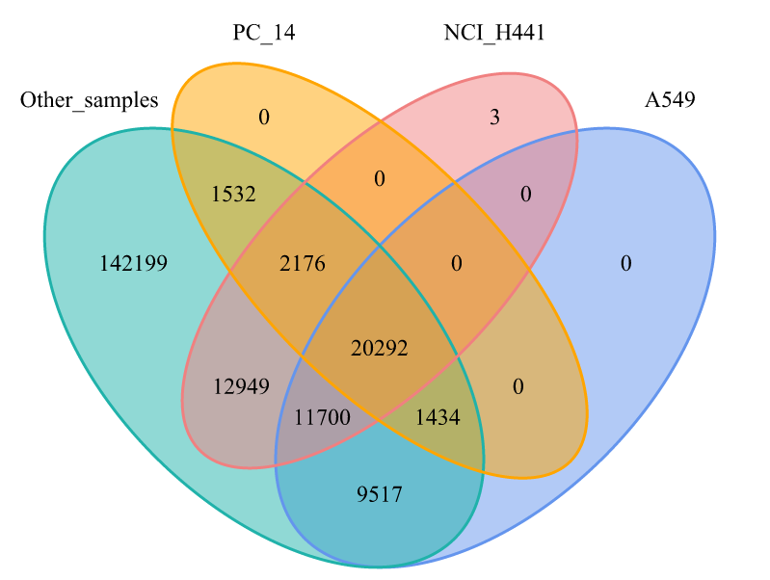
此外要用到的TSS-seq数据虽然是随机引物扩增,但是也经历过Poly(A)筛选,研究意义也大打折扣,最终课题只能作罢。既然课题成不了,又在上面浪费了不少时间,就写篇BLOG吐槽一下吧。
One More Thing, eRNA可能还有更要命的问题
这3篇文章的研究都是基于“eRNA是激活enhancer的标志”这个前提假设进行。但是后来看到2017年发表在Genome Biology上的一篇文章[5]通过数据分析对此提出了质疑:
Conclusions
Our results indicate that bidirectional transcription initiation from accessible chromatin is not sufficient for, nor specific to, enhancer activity. Transcription initiating at enhancers may be a frequent by-product of promiscuous RNA polymerase initiation at accessible chromatin and is unlikely to generally play a functional role in enhancer activity.
这才是跟风做研究最大的风险所在。有篇引用这篇文章的综述给这个起了个有趣的名字——“Validation Creep”[6]。很好奇过几年,这个方向会不会也像“心肌干细胞”一样最终被证伪呢?我会持续关注的。可能后面会单独开一篇BLOG来解读这篇文章(无责任挖坑)。
参考文献
- eRic数据库:https://hanlab.uth.edu/eRic/
- Andersson R, Gebhard C, Miguel-Escalada I, et al. An atlas of active enhancers across human cell types and tissues[J]. Nature, 2014, 507(7493): 455. https://www.nature.com/articles/nature12787
- Chen H, Li C, Peng X, et al. A pan-cancer analysis of enhancer expression in nearly 9000 patient samples[J]. Cell, 2018, 173(2): 386-399. e12. https://www.sciencedirect.com/science/article/pii/S0092867418303076
- Zhang Z, Lee J H, Ruan H, et al. Transcriptional landscape and clinical utility of enhancer RNAs for eRNA-targeted therapy in cancer[J]. Nature communications, 2019, 10. https://www.nature.com/articles/s41467-019-12543-5
- Young R S, Kumar Y, Bickmore W A, et al. Bidirectional transcription initiation marks accessible chromatin and is not specific to enhancers[J]. Genome biology, 2017, 18(1): 242. https://genomebiology.biomedcentral.com/articles/10.1186/s13059-017-1379-8
- Halfon M S. Studying transcriptional enhancers: the founder fallacy, validation creep, and other biases[J]. Trends in Genetics, 2018. https://www.sciencedirect.com/science/article/abs/pii/S0168952518302026
 作者:
作者:
果博东方客服开户联系方式【182-8836-2750—】?薇- cxs20250806】
果博东方公司客服电话联系方式【182-8836-2750—】?薇- cxs20250806】
果博东方开户流程【182-8836-2750—】?薇- cxs20250806】
果博东方客服怎么联系【182-8836-2750—】?薇- cxs20250806】
果博东方客服开户联系方式【182-8836-2750—】?薇- cxs20250806】
果博东方公司客服电话联系方式【182-8836-2750—】?薇- cxs20250806】
果博东方开户流程【182-8836-2750—】?薇- cxs20250806】
果博东方客服怎么联系【182-8836-2750—】?薇- cxs20250806】